问题分诊室
Cell:哺乳动物减数分裂重组图谱构建新方法:Ori-SSDS
发布时间: 2021-08-10
作者:十一月
有性生殖过程中会进行减数分裂,在一轮的DNA复制之后,再经过两次细胞分裂产生的单倍体配子。减数分裂过程中的遗传重组确保染色体的可靠分离,并建立了遗传连锁以及遗传模式。重组过程是由数百个程序化的DNA双链断裂(DNA double-strand breaks,DSBs)过程形成而引发的。在小鼠和人类中,DNA双链断裂别减数分裂特异性组蛋白甲基转移酶PRDM9所调控【1,2】。但是也有研究表明,个体中存在一个广泛的、不依赖于PRDM9的控制方式也会对减数分裂重组过程进行调控【3】。也就是说PRDM9蛋白定义了重组热点,然而,百万碱基级别的大规模重组模式独立于PRDM9。
为了揭开减数分裂过程中的重组模式,2021年7月13日,美国NIH遗传学与化学分院R. Daniel Camerini-Otero研究组发文题为Meiotic recombination mirrors patterns of germline replication in mice and humans,构建了一种用于定位哺乳动物细胞核组织中复制起点的新方法,从而可以将复制时间与全基因复制的模型相结合,揭开了对小鼠和人类生殖细胞复制模式以及减数分裂重组分布特征。

为了确定减数分裂复制的起点,作者们引用了一种叫做RNA引导新生短前导链(RNA-primed short nascent leading strands,SNS)的方法【4】进行测序。简单地说,RNA引物的前导链是通过使用lambda外切酶来消化缺乏RNA引物的DNA来进行分离的,冈崎片段虽然也有RNA引物,但是会因为片段大小这一点被选择性排除。为了适用于本工作的需要,作者们又对SNS-Seq的方法进行了优化,保留了新生链的方向性,将该技术称为Ori-SSDS(Origin-derived single-stranded DNA sequencing)【5】。Ori-SSDS的工作原理是将一个测序适配器(Adaptor)直接连接到了ssDNA,ssDNA自发地形成发夹环结构,双链DNA可以被处理,从而允许进行接头连接【6】(图1)。
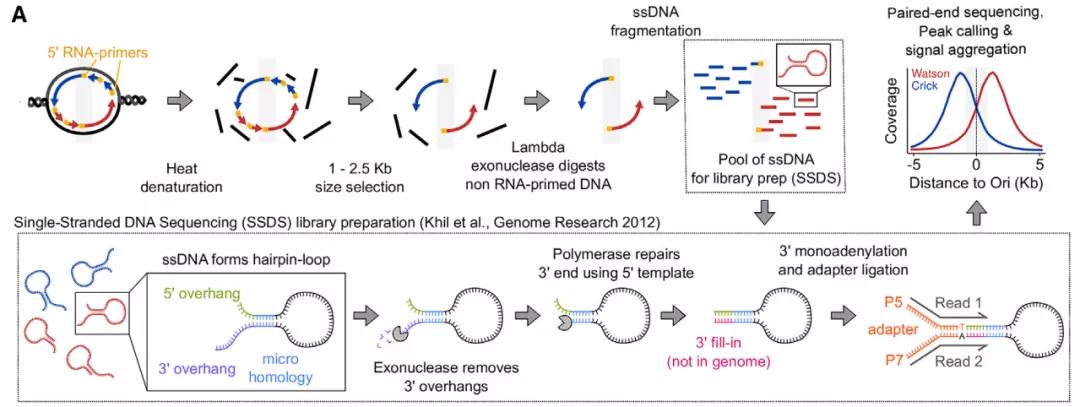
图1 Ori-SSDS工作原理
在从雄性小鼠个体的睾丸中进行的复制检测实验中,作者们检测到了11,209、12,406和13,476个复制起点,这表明该方法对于从个体动物中绘制复制起点是足够敏感的。而在对照实验中,作者们使用不可DNA复制的组织比如精子或者去除RNA引物的前导链复制起点进行检测。该结果进一步说明了Ori-SSDS测序方法的准确性与有效性。另外通过在培养的细胞中进行Ori-SSDS测序,作者们确认无论是从组织还是从培养的细胞中均可以从全基因组中识别复制的起点。
进一步地,作者们对复制起点区域的特征进行总结。从小鼠睾丸中鉴定出的复制起点,作者们发现在基因组中分布是不均匀的,并且倾向于聚集在基因丰富的区域,作者们还发现其中66%的复制起点发生在转录起始位点(Transcription start site)的1kb以内的位置。位于起点中心的DNA是相对柔性的而两侧的DNA是相对刚性的,这反映了ORC复合物对于弯曲DNA的要求。另外,DNA复制起点区域富含GC,GC含量、CpG密度、GpC密度均与起始效率呈正相关的特点。68%的复制起点于染色质可及性中CpG岛相一致。但是不可及的染色质区域中的复制起点会比较难以预测。祖宗们在复制起点并未发现任何保守的序列特征基序,但是可以用CpG的密度可以作为预测复制起点的特征点。因此,总结来说,生殖细胞中的复制起点主要在靠近启动子、开放的染色质以及富含CpG岛的区域。
由于复制起点分布命运的不均匀,因此由早期和晚期不同基因组复制部分的组成。为此,作者们希望对减数分裂过程的复制时间进行检测。作者们通过细胞分选将表达分裂期细胞标记物SYCP3的细胞进行分选,并对复制时间进行测序(Replication timing-Seq,RT-Seq)。早期和晚期复制区域可以通过高和低复制起点密度所定义,这与最近在其他哺乳动物细胞类型中的研究结果是相一致的【7】。
作者们想知道复制时间是否可以作为复制特征,为此作者们检查了S期极早期、早期、中期以及晚期最佳拟合的模型运行时间。作者们发现降低复制起点会延长S期复制阶段。进一步地,作者们想知道DNA复制与重组过程之间的关系。在减数分裂中,DNA复制后发生程序化的DSB形成。减数分裂DSB会通过一个级联过程被检测到,PRDM9作为甲基转移酶三甲基化H3K4和/或H3K36,从而招募DSB复合物。作者们发现所有重组过程都富集在早期复制区域(Early replicating regions,ERRs)。因此,该结果确认减数分裂重组过程在时间和空间上与DNA复制存在相关性。
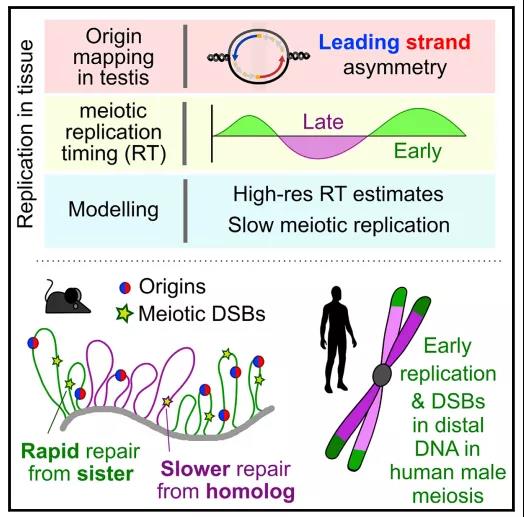
图2 工作模型
总的来说,作者们通过对将一种定位哺乳动物细胞和组织中复制起源的新方法与复制时间以及全基因组复制的计算模型相结合的方法Ori-SSDS,构建了雄性减数分裂过程中复制起点的差异可以用于预测小鼠和人类减数分裂期间的重组图谱。
原文链接:
#
参考文献
1 Baudat, F. et al. PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice. Science (New York, N.Y.) 327, 836-840, doi:10.1126/science.1183439 (2010).
2 Myers, S., Bottolo, L., Freeman, C., McVean, G. & Donnelly, P. A fine-scale map of recombination rates and hotspots across the human genome. Science (New York, N.Y.) 310, 321-324, doi:10.1126/science.1117196 (2005).
3 Davies, B. et al. Re-engineering the zinc fingers of PRDM9 reverses hybrid sterility in mice. Nature 530, 171-176, doi:10.1038/nature16931 (2016).
4 Bielinsky, A. K. & Gerbi, S. A. Discrete start sites for DNA synthesis in the yeast ARS1 origin. Science (New York, N.Y.) 279, 95-98, doi:10.1126/science.279.5347.95 (1998).
5 Cayrou, C., Grégoire, D., Coulombe, P., Danis, E. & Méchali, M. Genome-scale identification of active DNA replication origins. Methods (San Diego, Calif.) 57, 158-164, doi:10.1016/j.ymeth.2012.06.015 (2012).
6 Khil, P. P., Smagulova, F., Brick, K. M., Camerini-Otero, R. D. & Petukhova, G. V. Sensitive mapping of recombination hotspots using sequencing-based detection of ssDNA. Genome research 22, 957-965, doi:10.1101/gr.130583.111 (2012).
7 Dileep, V. & Gilbert, D. M. Single-cell replication profiling to measure stochastic variation in mammalian replication timing. Nature communications 9, 427, doi:10.1038/s41467-017-02800-w (2018).