在群体遗传结构研究过程中,主成分分析(Principal Component Analysis,即PCA)、进化树分析、群体结构、LD连锁不平衡分析是比较常见的分析方法。多种分析方法结合,可以帮助我们探索群体遗传结构、遗传多样性,推断群体进化过程。此处我们基于示例数据进行群体遗传结构的分析及结果解读。
分析内容及使用软件:
· PCA分析 :利用GCTA进行PCA分析,关于PCA分析具体步骤之前有详细介绍,此处不再赘述。
· 进化树分析:使用Plink构建IBS遗传距离矩阵,以此为基础,利用MegaX构建进化树,进化树美化使用在线网站iTOL。
· 群体结构分析:利用Admixture软件进行群体结构分析。
· 连锁不平衡分析:利用PopLDdecay软件进行连锁不平衡分析。
本次示例数据来源于3个品种,共220个样本, 分析之前需要准备:
· 基因型数据(plink格式为例);
· 标识3个品种分组信息的文件,第一列样本名称,第二列组别。
目的:通过基因型数据,探索待测220个样本3个品种间的遗传联系。
基因型数据质控
使用Plink(V1.90)软件进行质控,保留分型质量最好的位点进行后续分析,命令行如下。
plink --file plink --geno 0.1 --maf 0.01 --recode --out bestqc主成分分析
通过PCA分析可以大致了解群体的遗传结构,在PCA图上,空间直线距离越近的个体遗传关系越近,越远的个体遗传关系也越远,但PCA只能提供一个比较宏观的个体关系。
常用软件:PLINK、GCTA,此处以GCTA为例,命令行如下。
gcta_1.94.0beta/gcta64 --bfile bestqc --autosome-num 50 --make-grm --make-grm-alg 0 --out kinship0gcta_1.94.0beta/gcta64 --grm kinship0 --pca 3 --out gcta
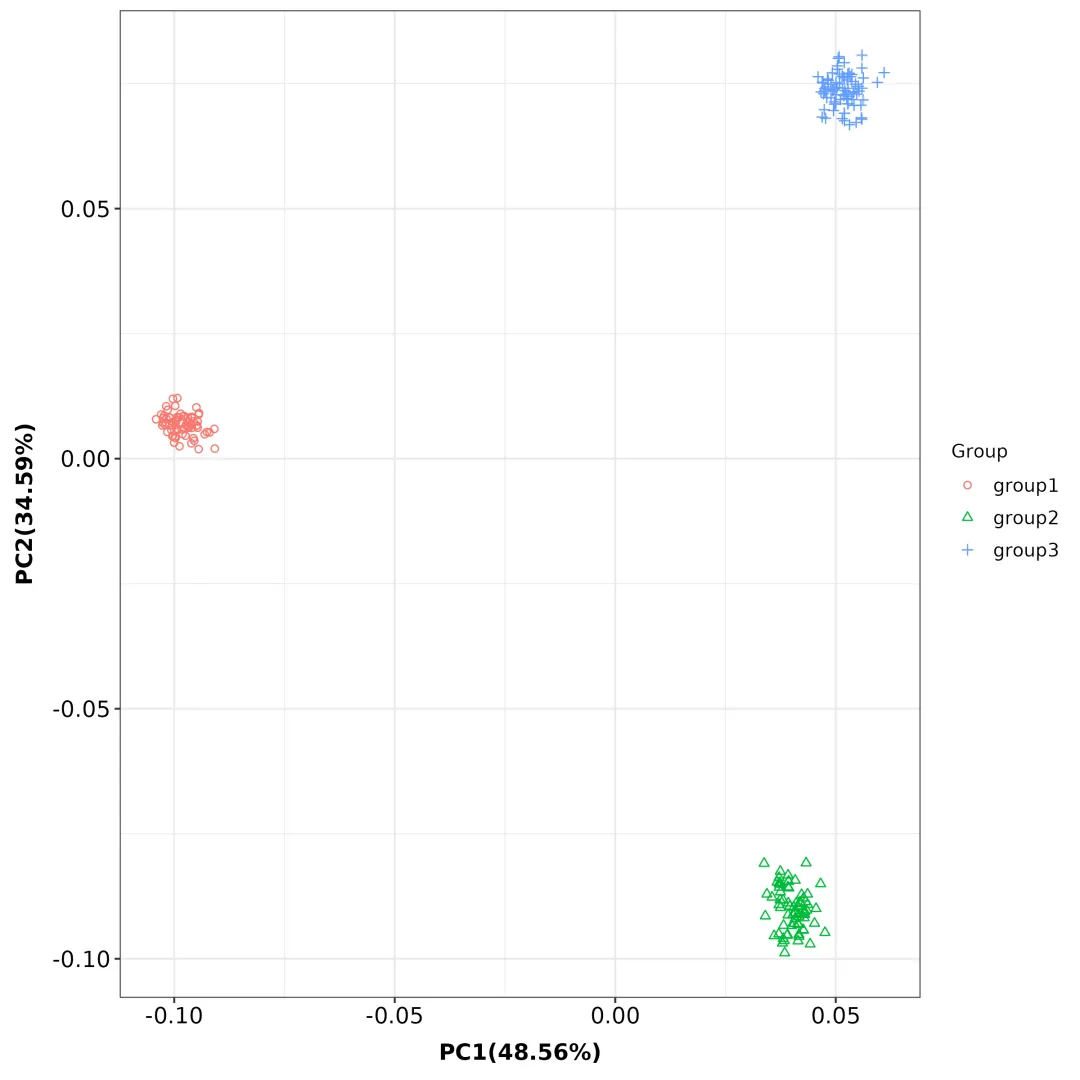
图1 PCA可视化结果
结果解读:通过PCA分析结果,可以探索待测样本分群信息,观察群体分层、亚群之间的遗传差异。
如示例,图中不同颜色代表已知的3个分组,PCA分析结果可见220个分析样本呈明显的群体分层,大概分成3个群体,与已知分组一致。
进化树分析
系统发育树是描述群体间分化顺序的分支图或树,用来表示群体间的进化关系。从进化树结果中,我们可以大致推断哪些样本的遗传特征较为接近。
构建进化树方法有多种,如邻接法(Neighbor-Joining, NJ)、最大似然法(ML, Maximum likelihood)等,此处以邻接法为例,命令行如下。
# Step1基于Plink构建IBS遗传距离矩阵plink --bfile bestqc --distance 1-ibs square flat-missing --genome --out IBSmatrix --chr-set 50# Step2整理为.meg输入格式,基于MEGA构建进化树megacc -a infer_NJ_distances.mao -d ibs_dismatrix.meg -o mega_IBSout# 指定分析文件: infer_NJ_distances.mao,此处指定为邻接法,此文件来源于MegaX 软件导出;除以上外也可直接用windows版MEGA软件。# Step3 进化树美化:一般建树后导出格式为 *.nwk 的文件,导入到相关软件如在线网站ITOL绘制进化树,或者基于R包ggtree进行修图调整。
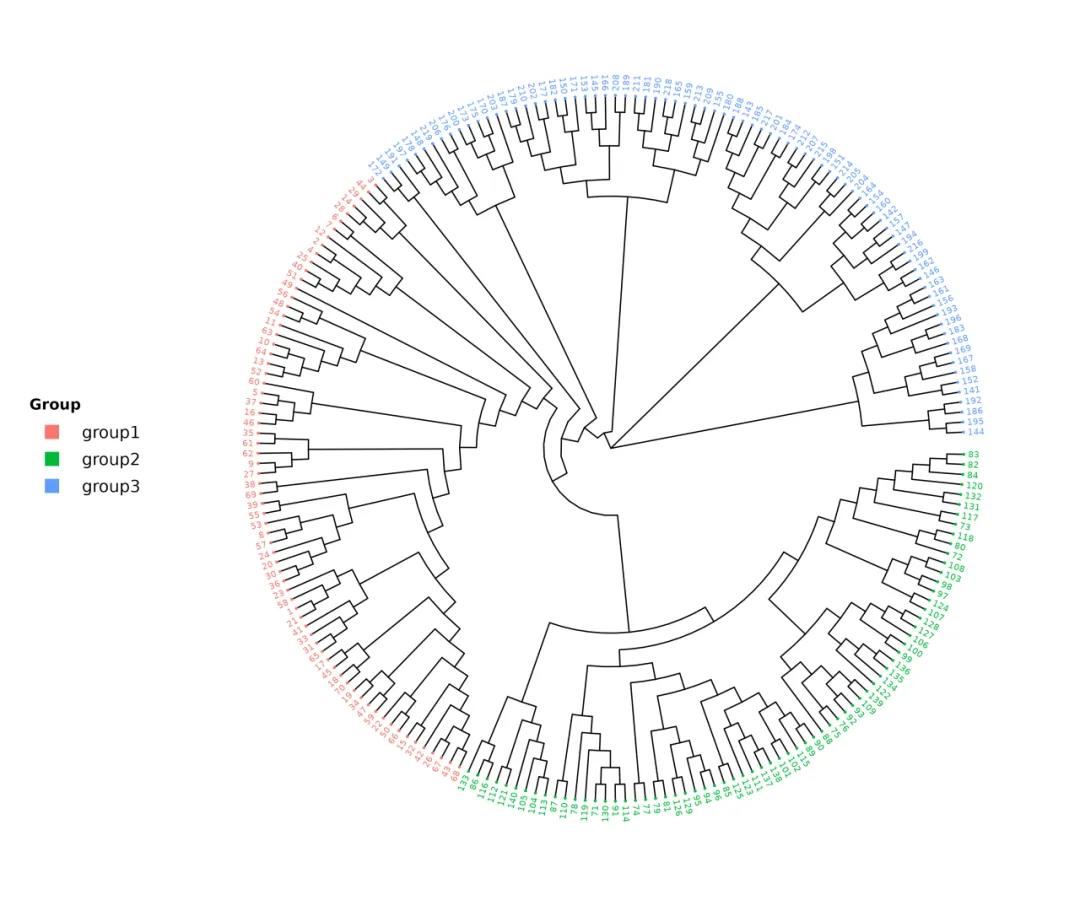
图2 进化树可视化结果
结果解读:进化树可以直观看到各样本间的亲缘关系,分析样本量大、样本间差异较大时,可用圈图的形式展示。
如示例,图中添加已知分组信息,以便观察样本分布情况,不同颜色代表已知的3个分组。进化树结果与PCA分析结果一致,220个分析样本呈明显的群体分层,图中同一个颜色就表示这些树枝所代表的样本是属于同一个品种,与已知品种分组一致,3个群体间相对独立。
群体结构分析
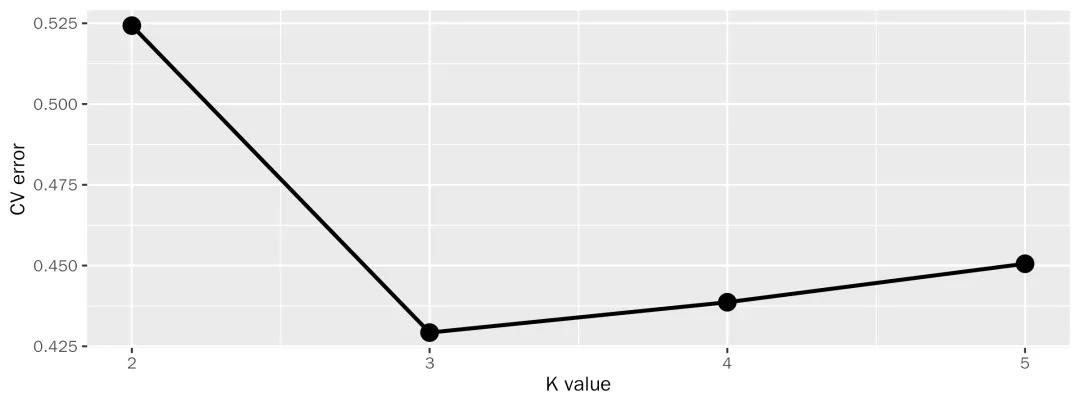
群体遗传结构分析,也称为祖源成分分析,有助于理解进化过程,可以通过基因型和表型的关联研究确定个体所属的亚群。根据给定的群体信息取多个K值,并利用Admixture软件进行群体结构分析。一般CV值越小所对应的K值越佳,以此确定最佳分群数。
群体结构分析软件常见为Structure、Admixture,其中Admixture运算速度相对较快,此处以Admixture为例,命令行如下。
# Step1 Admixture分析for K in 2 3 4 5; do admixture --cv bestqc.bed $K | tee log${K}.out; done# Step2 获取最佳K值grep -h CV log*.out# Step3 可视化Admixrture运行结束.Q后缀文件用于画图,但无样本名称,可从.fam提取样本名,与Q矩阵合并。用堆叠柱状图、或参考R包pophelper进行可视化。
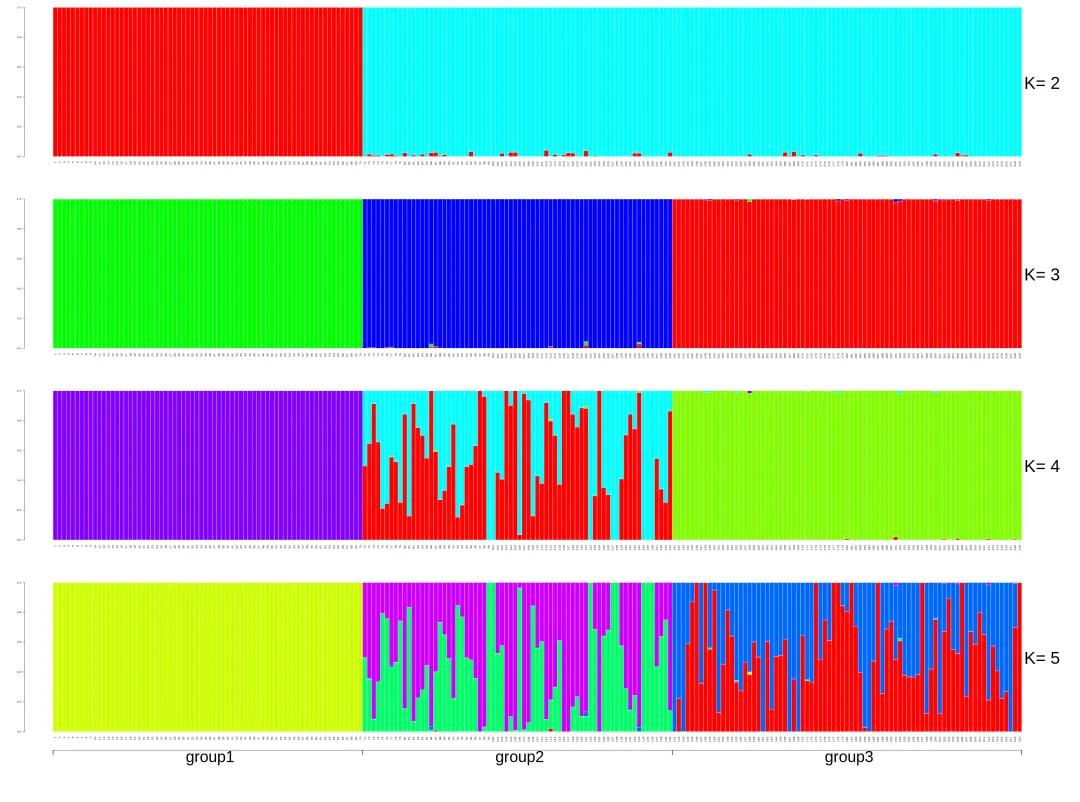
图3 群体结构分析结果
结果解读:群体遗传结构分析,除可以推测最佳分群数、探索群体间基因交流,还可以推测出单个样本混血程度。
如示例,右侧K值表示,本次研究假设祖先群体个数为2到5,其中不同颜色片段的长度表示该个体基因组中某个祖先或亚群所占的比例,横坐标表明已知样本所属分组群体名称。通过颜色及某个祖先成分占比,可以对种群中的个体进行不同亚群的划分。
当K=2时,所有样本可大致分为2个亚群,group1样本可划分为一个亚群,group2、group3可划分为一个亚群;当K=3时,220个个体明显分为3 个群体,来自group2的个体与group3的个体分开,血统较为纯正;当K=4时,来自group2的个体内部分开;当K=5时,来自group3的个体内部分开,其中的group2、group3个体基因组中包含多个祖先成分,这可能是杂交基因渗入等导致群体内呈现遗传多样性。
同时,K=3时,CV值越小,为待测样本最佳分群数,该测试种群中的个体可以划分成3个亚群,最佳分群数与目标品种数一致。
连锁不平衡
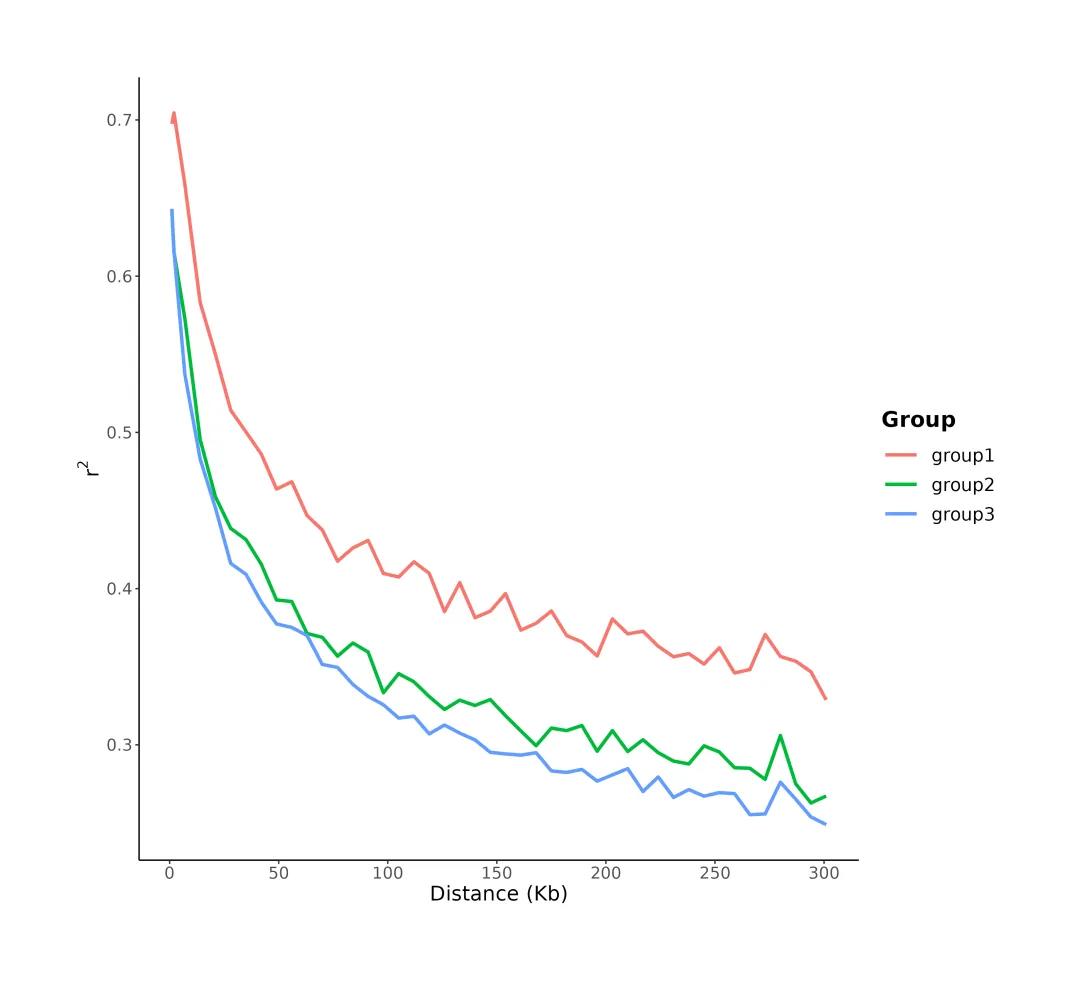
连锁不平衡(Linkage disequilibrium, LD)是群体遗传学研究中常见的分析内容,当位于同一条染色体的两个等位基因(A,B)同时存在的概率大于群体中因随机分布而同时出现的概率时,就称这两个点处于LD状态,通常用D’和r2值表示,命令行如下。
# 计算LD DecayPopLDdecay -InVCF bestqc.vcf -MaxDist 300 -OutStat out
图4 LD衰减图
结果解读:LD分析可以揭示不同品种群体在进化过程中受到选择的强度及遗传物质多态性的高低,通常LD衰减速度越慢,表明其受选择程度越强。
如示例,横坐标表示物理距离,单位为Kb,纵坐标用r2的值表示平均连锁不平衡程度。group3群体LD衰减速度最快,由此推测该群体受选择程度较弱。group1群体LD衰减速度最慢,由此推断该群体受选择程度较强。
通过PCA分析、进化树、群体结构、连锁不平衡分析可以大致了解群体的遗传结构,以上结果相互之间可以进行验证,常被用来推断群体分层、进化关系等。本文以待测220个样本,基于基因型数据利用以上分析方法综合判断:220个样本最佳分为3个群体,各群体间遗传距离较远,各群体内样本聚集、遗传背景较一致。
参考文章:
[1] Purcell et al. 2007 PLINK: a tool set for whole-genome association and population-based linkage analyses.
[2] VanRaden 2008 Efficient Methods to Compute Genomic Predictions.
[3] Yang et al. 2011 GCTA: a tool for Genome-wide Complex Trait Analysis.
[4] kumar et al. 2018 MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms.
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司








 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号