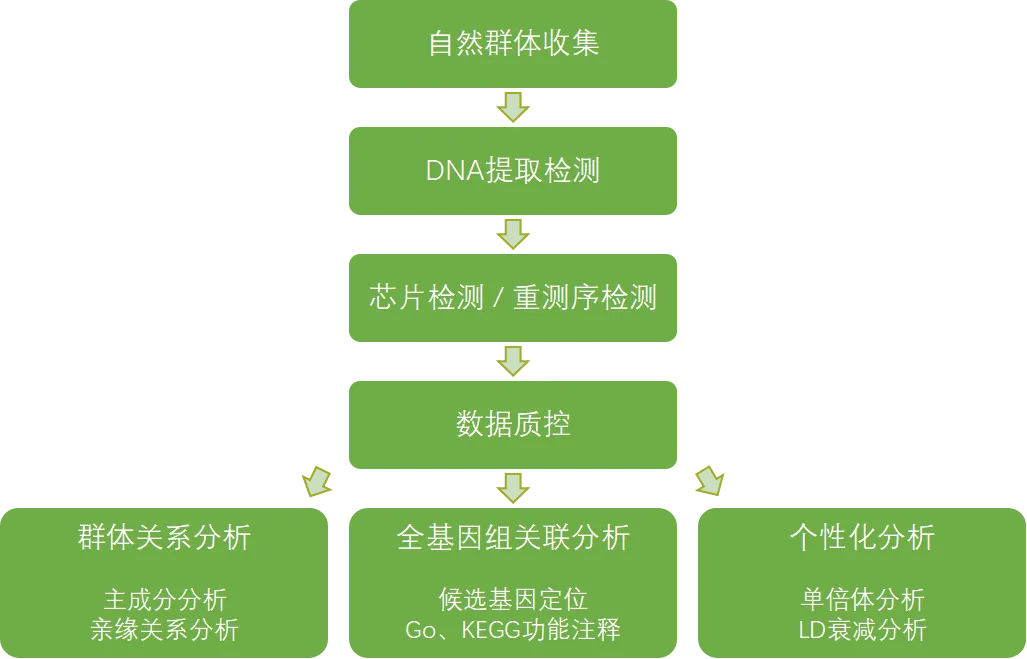
近年来,常规育种辅助分子标记大大加速育种进程,在农作物与畜禽的育种或品种改良工作中广泛应用。在育种工作中,以分子标记为辅助手段,进行早期筛选,可以大大提高杂交后代轮回亲本的恢复速度。通过选择最适合的亲本进行杂交与高效快速精准的分子选择,聚合各种重要农艺性状的优异基因,从而高效培育农艺性状优异新品种的先进技术体系,大大提高育种效率。
基因芯片应用和推广使育种工作向着低成本、高通量和高质量的方向不断发展,康普森以先进的设计流程、严谨的开发指标、成熟的检测技术助力开发了多款基因育种芯片,基于前期群体样本重测序数据,利用BSA、QTL定位、GWAS分析、选择信号等的方法挖掘多种重要经济性状关联位点,转化为功能基因芯片,为分子育种工作提供有效的基因分型工具。
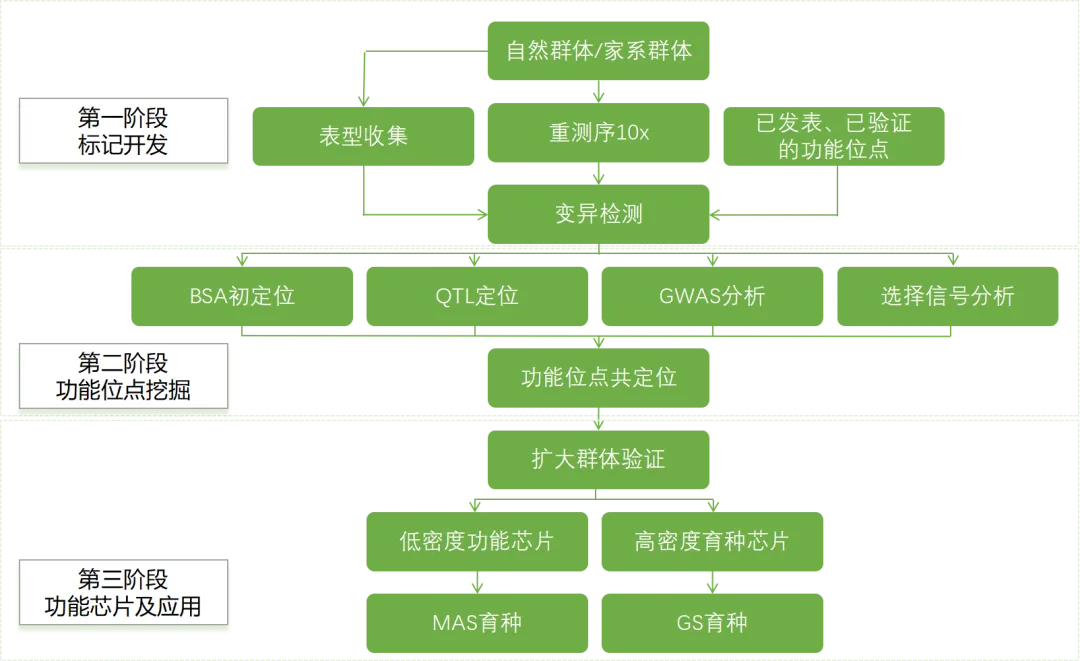
图1 功能芯片定制方案流程图
BSA (Bulk Segregant Analysis, BSA)是快速鉴定与目标性状紧密连锁的分子标记的方法。针对该家系目标性状表型极端的子代分 别混合成的两个样本池进行测序,同时对亲本进行测序,检测与性状相关联的位点并注释, 研究基因控制目标性状的机制,是一种快速、准确、性价比高的定位方法。
技术策略
样本类型:家系群体(F2、RILS、DH家系群体等)
样本数:常规建议2个亲本+2个极端子代混池
基因型检测:亲本10x重测序、子代20-30x测序
适用性状:质量性状、数量性状、突变性状,单个性状定位,表型越极端越好
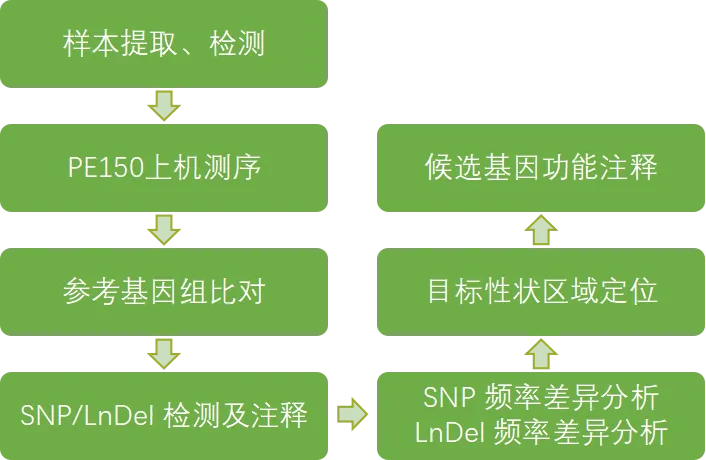
技术路线
文献案例: High-resolution bulked segregant analysis enables candidate gene identification for bacterial wilt resistance in Italian ryegrass (Lolium multiflorum Lam.) [1]
物种:黑麦草
分型策略:重测序
发表时间:2024年
期刊:The Plant Journal
IF:7.2(2024年)
本研究通过Xtg-ART群体衍生的F2群体,对抗病池和感病池进行高深度全基因组测序,使用BSA的方法(ΔSNP index、G和ED)确定了多花黑麦草一个300kb的基因组区域对细菌枯萎病具有抗性,最终定位到4号染色体上,位于M2289_v2基因组28800000bp-28900000bp区间,获得12个抗病相关的候选基因。
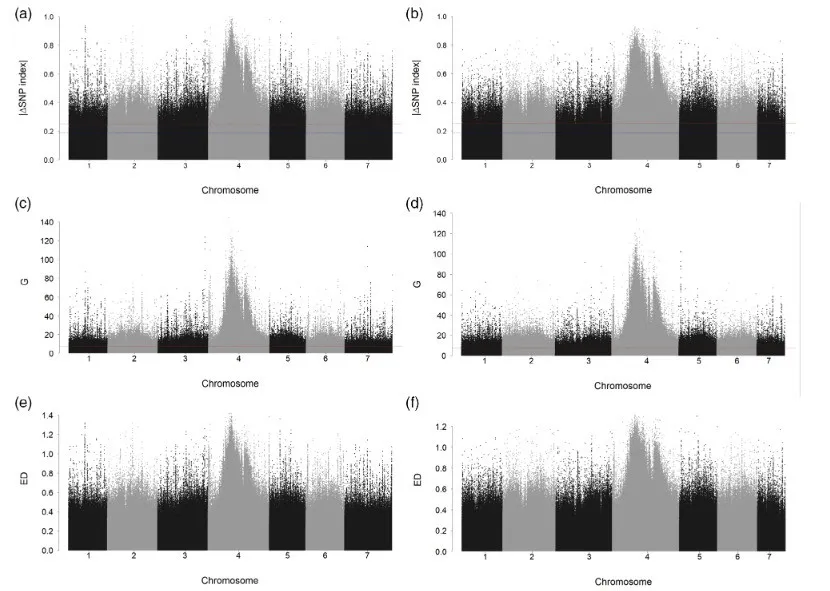
图2 细菌枯萎病BSA定位结果
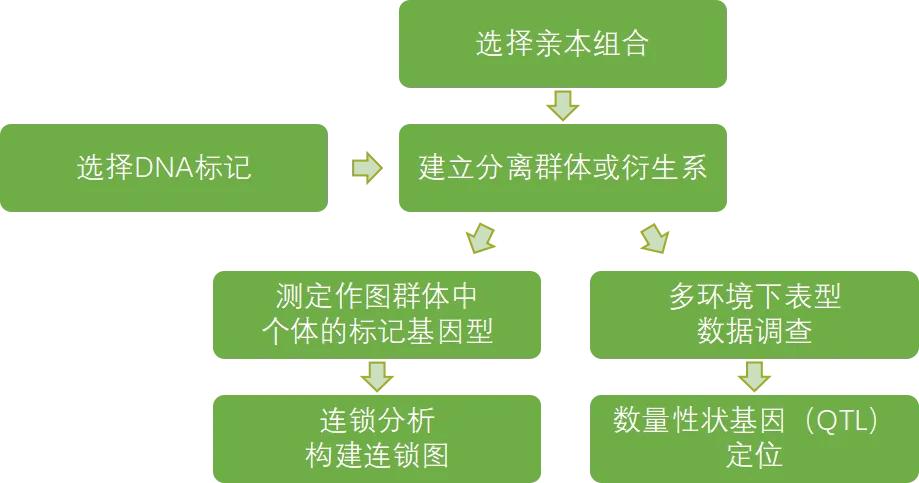
遗传图谱也叫连锁遗传图谱(Genetic linkage map),是以基因或者遗传标记之间的连锁和重组交换值构建的图谱。QTL定位是通过对遗传标记与性状之间相关性的分析,将一个或多个QTL定位到染色体的遗传标记之间。
技术策略
样本类型:家系群体(F2、RILS、DH、BC群体等)
样本数:150以上
基因型检测:重测序、基因芯片
适用性状:质量性状+数量性状,多个性状定位
技术路线
文献案例:Identification of genes for drought resistance and prediction of gene candidates in soybean seedlings based on linkage and association mapping[2]
物种:大豆
分型策略:重测序
发表时间:2022年
期刊:The Crop Journal
IF:6.6(2022年)
利用大豆的234个重组自交系群体(RIL)、259个大豆品种组成的自然群体进行干旱胁迫处理,采集两个环境下干旱胁迫表型数据。基于重测序数据开发的SNP标记构建的遗传图谱,定位到18个抗旱相关的QTLs,全基因组关联分析获得69个抗旱显著关联SNP。通过连锁分析和关联分析的共定位发现,两个显著关联的SNP位于两个QTL(qPH7-4和qPH7-6)置信区间内,对显著关联的SNP ss246200757和ss246238768两侧延伸300 kb进行LD分析,发现了3个block。
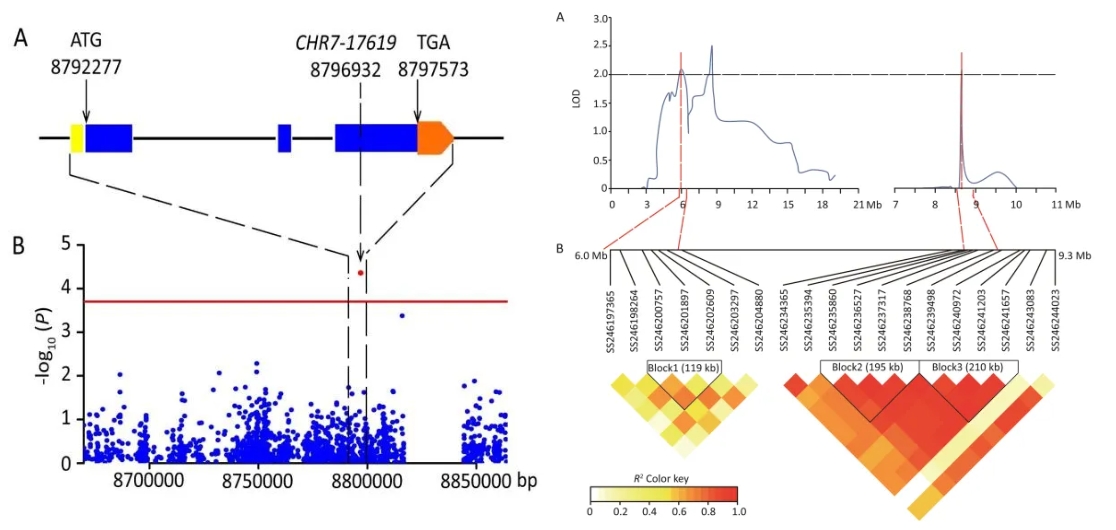
图3 抗旱性状定位结果
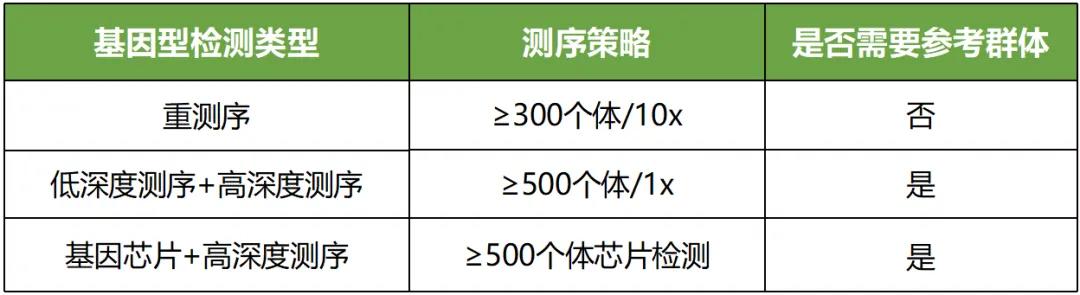
全基因组关联分析(Genome-wide association study,GWAS)是一种对全基因组范围内的常见遗传变异(单核苷酸多态性 SNP)基因总体关联分析的方法,可直接鉴定出与表型变异显著相关且具有特定功能的基因位点或标记位点,适用于挖掘优良性状等研究。
技术策略
样本类型:自然群体、育种群体
样本数:建议300以上
基因型检测:常规推荐10x重测序、LcWGS低深度重测序、基因芯片
技术路线
文献案例:Accurate haplotype construction and detection of selection signatures enabled by high quality pig genome sequences[3]
物种:猪
分型策略:1096个重测序+1490个芯片检测
发表时间:2023年
期刊:Nature Communications
IF:16.6(2023年)
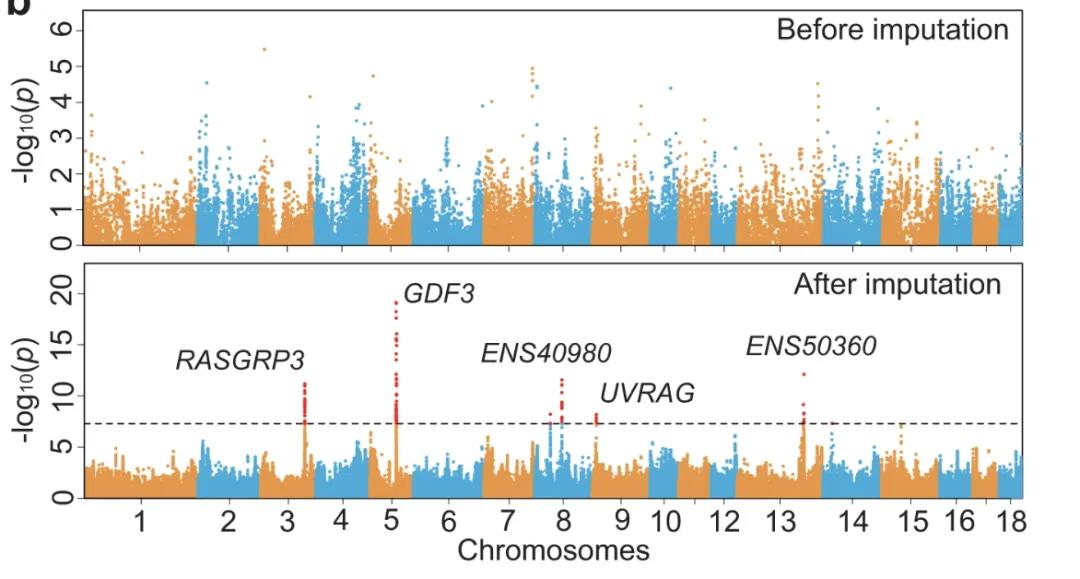
对全球43个猪种共计1096个高深度重测序样本,平均测序深度17x,结合系谱信息、有效单倍型读长以及连锁不平衡信息,构建了覆盖全球多品种、大样本、最准确的千猪单倍型数据库,展示了猪复杂性状因果机制解析、基因组选育以及进化选择方面的应用。基于构建的单倍型参考面板,将50k芯片基因型数填充至千万级别,利用填充后的基因型进行GWAS分析,与芯片数据相比新鉴定到5个与肌内脂肪含量显著关联位点,育种值估计与芯片估计相比准确性提升了36.7%。
图4 填充前后的肌内脂肪含量(IMF)关联分析结果
功能芯片开发
基于前期性状定位挖掘到的功能标记以及基因组重要区域的背景标记,开发一款适用于大量群体样本检测的低密度功能芯片,高效、快速、低成本实现基因分型,不仅可以提高分子育种选择的准确性,明确目标基因导入情况,加速遗传背景回复纯化,也为进一步精准育种和更多优良基因的聚合做准备。
案例1-中豆芯二号-大豆功能芯片
中国农业科学院作物科学研究所与北京康普森生物技术有限公司合作,依托靶向测序技术研发了一款基因型鉴定芯片 ——“中豆芯二号”,是一款低密度的大豆育种芯片,包含 5500 个位点。涵盖大量高产优质的克隆基因,最新的功能位点、关联及连锁分析的研究成果,结合中豆芯一号优选位点定制而成,可广泛应用于育种材料遗传评估、回交育种、聚合育种,F1真假杂交种鉴定,品种鉴定等。
图5 中豆芯二号育种芯片
案例2-骆驼1k功能芯片
阿拉善盟畜牧研究所道勒玛老师联合内蒙古农业大学郭丽丽博士以及北京康普森农业科技有限公司,共同完成了“驼铃一号”双峰驼1k功能位点液相育种芯片的研发。该芯片的研发为阿拉善乳用驼选育提供了更高效的新工具,结合多个关键产乳性状(乳产量、乳蛋白含量、乳脂含量、乳糖含量等),鉴定显著相关的SNP位点,采用康普森CAGT®靶向捕获测序技术,最终设计完成此包含1004个SNP位点的靶向捕获芯片。
图6 驼铃一号
参考文献
Florian G, Yutang C, Verena K, et al. High-resolution bulked segregant analysis enables candidate gene identification for bacterial wilt resistance in Italian ryegrass (Lolium multiflorum Lam.). Plant J. Published online February 27, 2024. doi:10.1111/tpj.16693.
Yanjun Zhang, Zhangxiong Liu, et al.Identification of genes for drought resistance and prediction of gene candidates in soybean seedlings based on linkage and association mapping.The Crop Journal.10.3.2022,https://doi.org/10.1016/j.cj.2021.07.010.
Tong, X., Chen, D., Hu, J. et al. Accurate haplotype construction and detection of selection signatures enabled by high quality pig genome sequences. Nat Commun 14, 5126 (2023). https://doi.org/10.1038/s41467-023-40434-3.
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司








 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号