主成分分析(Principal Component Analysis,即PCA),是一种数据降维技巧,从高维度数据中提取关键的信息,以便使用较少的变量就可以对样本进行有效区分,此处我们基于2款常用软件进行PCA分析。
使用软件
Plink :这是一个生信分析使用频率相对比较高的软件,功能很强大,具体功能请参考官网,此处我们基于该软件进行PCA分析。
软件官网:https://www.cog-genomics.org/plink/1.9/credits
GCTA:软件全称 Genome-wide Complex Trait Analysis,最初用于全基因组复杂性状分析,目前已扩展到许多数据分析。此处我们基于该软件进行亲缘关系分析,进而进行PCA分析。
软件官网:https://yanglab.westlake.edu.cn/software/gcta/
R语言:基于ggplot2包进行PCA结果可视化。
本次示例数据来源于3个品种,共224个样本。PCA分析之前,我们需要准备:1. 基因型数据;2. 如果有关注品种或采样地等其它信息,则需要准备一个标识分组信息的文件。
基因型数据:此处基因型数据以plink格式文件为例,含map 、ped文件。map 文件主要是储存位点信息,每行代表一个位点,该文件含染色体、SNP位点名称、摩尔根距离、物理位置信息。ped 文件主要是储存每个样本的基因型信息,每行代表一个样本,含:家系 ID、个体 ID、父本 ID、母本 ID、性别、性状、样本的分型信息(每两列为一个 SNP)。
分组信息:如果有品种/采样地等其它信息,可以准备分组文件group.csv。含样本名称及组别。

基因型数据质控
主成分分析之前需要对基因型数据进行基本质控,使用Plink(V1.90)软件对样本检出率、SNP位点进行质控,保留分型质量最好的位点进行后续分析,命令行如下。
plink --file plink --geno 0.1 --maf 0.01 --recode --out bestqc
主成分分析
此处列举了2种PCA分析软件,具体如下:
方法一:基于plink软件进行PCA分析
# 基于 plink 进行 PCA
plink --file bestqc --pca 224 header
# 可视化
library(ggplot2)
dat=read.table(file="plink.eigenvec",header=T,as.is=T)[,1:5]
group=read.csv(file="group.csv",header=T,as.is=T)
dat$group=group$breed
plot=ggplot(dat,aes(x=PC1,y=PC2)) +
geom_point(size=3,aes(color=group))
ggsave(file="PCA_1_2.png",plot,width=6,height=6,device="png")
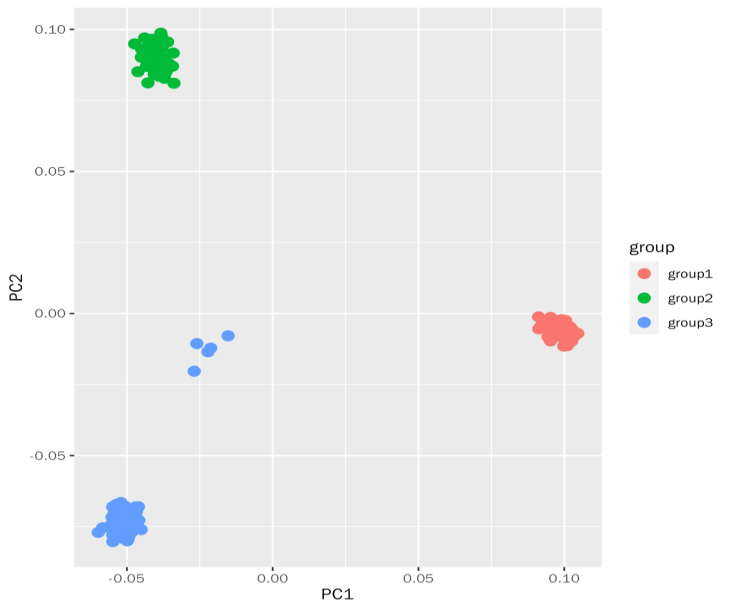
可视化结果如图所示:空间直线距离越近的个体遗传关系越近,越远的个体遗传关系也越远。基于plink软件进行PCA分析,结果来源于3个品种的224个样本出现明显的群体分层。
图1 基于plink软件PCA结果
方法二:基于GCTA软件进行PCA分析
gcta_1.94.0beta/gcta64 --bfile bestqc --autosome-num 50 --make-grm --make-grm-alg 0 --out kinship0
gcta_1.94.0beta/gcta64 --grm kinship0 --pca 3 --out gcta
# 可视化
library(ggplot2)
dat=read.table(file="gcta.eigenvec",header=F,as.is=T)[,1:5]
colnames(dat)=c("FID","IID","PC1","PC2","PC3")
group=read.csv(file="group.csv",header=T,as.is=T)
dat$group=group$breed
plot=ggplot(dat,aes(x=PC1,y=PC2)) +
geom_point(size=3,aes(color=group))
ggsave(file="PCA_1_2.png",plot,width=6,height=6,device="png")
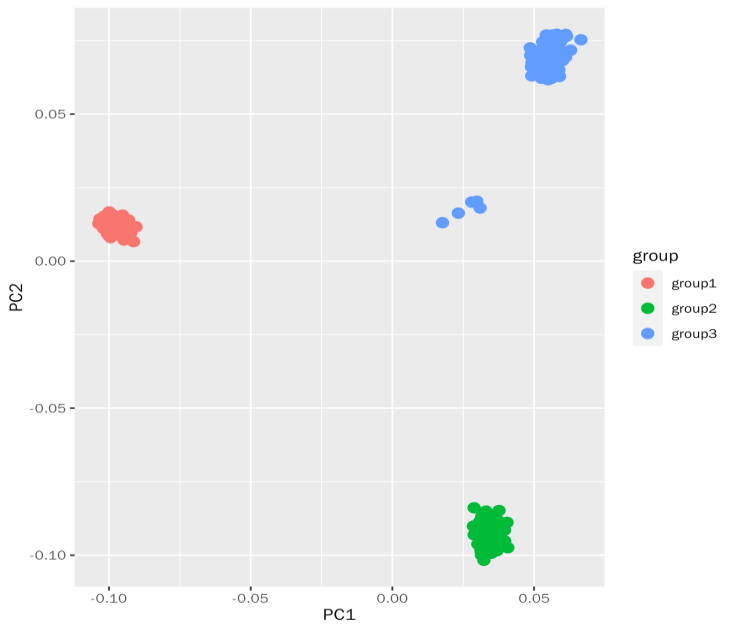
可视化结果如图所示,同样出现明显的群体分层。
图2 基于GCTA软件PCA结果
由上可见,GCTA软件需要先手动进行个体间亲缘关系的计算,再进行PCA分析。其中构建亲缘关系矩阵有2种方法可以选择,第一种:Yang的方法,参数 --make-grm 0 ;第二种:VanRaden的方法,参数 --make-grm-alg 1。
而plink软件通过参数 --pca 即可一步进行PCA分析。同时也可分步进行,基于plink软件先构建亲缘关系矩阵(参数使用 --make-rel ,软件官网说明默认使用Yang方法),再基于GCTA软件进行PCA分析。
应用示例
PCA分析应用广泛,例如1. 推断群体分层、进化关系,与进化树,群体结构、连锁不平衡分析结果互相验证。2. 检查离群样本,有选择地剔除离群样本(如取样错误、污染)。3. 生物学重复评估,判断哪些样本之间的遗传相似性较高。
示例一:辅助推断群体分层、进化关系。
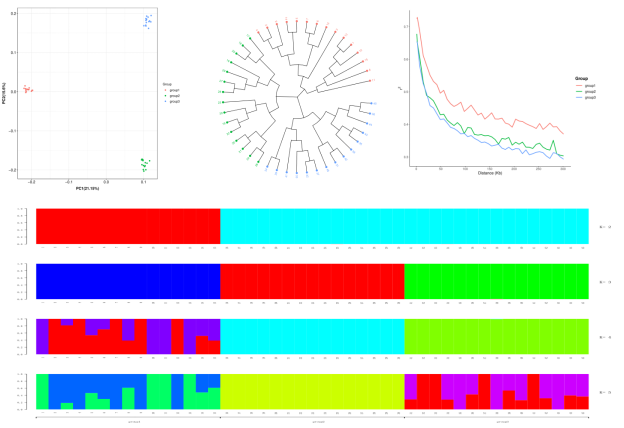
图3 群体遗传分析结果
说明:
1. 从PCA和进化树的结果可以看出,3个群体间遗传距离较远、遗传背景较独立。
2. 从LD衰减结果可以看出,group3群体LD衰减速度最快,由此推测该群体受选择程度较弱;group1群体LD衰减速度最慢,由此推断该群体受选择程度较强。
3. 从群体结构结果可以看出,所有样本可分成3个亚群,与分析品种数一致。
示例二:辅助推断疑似离群样本
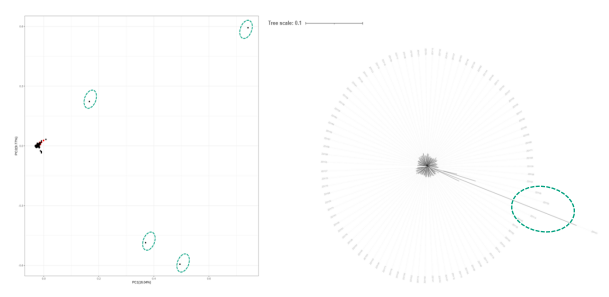
图4 PCA、进化树分析结果
说明:通过PCA和进化树可以大致了解群体的遗传结构。示例数据来源于1个品种,结合PCA、进化树可以初步推断绿色框选的4个样本,疑似为该品种中的离群样本。
参考文献
[1] Purcell et al. 2007 PLINK: a tool set for whole-genome association and population-based linkage analyses.
[2] VanRaden 2008 Efficient Methods to Compute Genomic Predictions.
[3] Yang et al. 2011 GCTA: a tool for Genome-wide Complex Trait Analysis.
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司







 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号