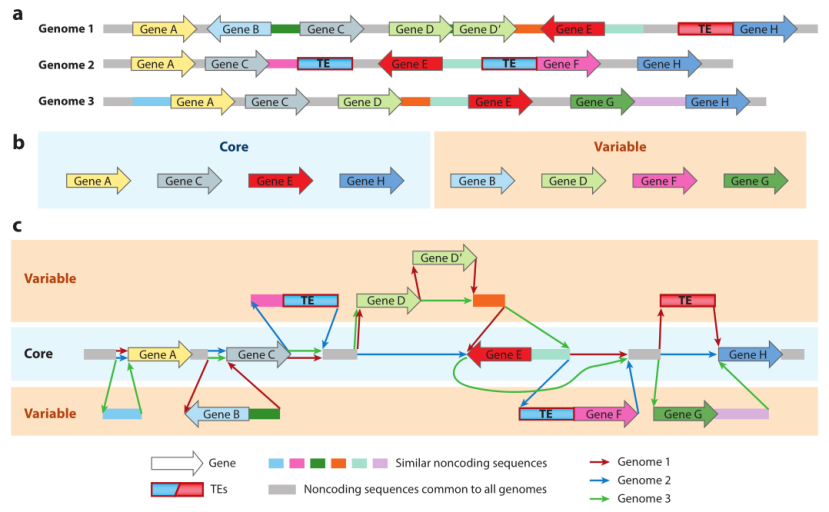
随着研究人员对生物基因组的深度解析,大家逐渐认识到任何一个物种单一基因组不能完全代表该物种内的遗传多样性,不同品种/品系在进化过程中基因组中出现的广泛的遗传变异,尤其是大尺度的结构变异(Structural variation,SV)扩大了它们间的序列差异。为了突破这一局限,完整捕获和解析群体中所有的遗传信息,科学家提出了泛基因组(Pan-genome)的概念,以功能基因为泛基因组的基本元素,泛基因组是指群体内所有功能基因的总和,通过泛基因组研究群体内功能基因的分布和结构变异,捕获和呈现群体内全部的功能基因。从基因组结构性来看,泛基因组是通过对群体内的基因组序列进行比较分析,收集和呈现群体内全部基因组序列,并以序列变异作为泛基因组分析的基本元素[1]。泛基因组可以分为核心基因组(core genome)和可变基因组(variable genome)(图1)。核心基因组是指存在于所有个体中的基因(序列),可变基因组是指只存在于部分个体中的基因(序列)。从进化的角度看,核心基因组的基因主要包括维持该物种存活所必需的最基本生理功能的基因,这些基因在细胞内行使极其关键的功能。可变基因组则包含了群体内全部的可变基因。研究发现,很多物种都存在大量非必需基因组,非必需基因很可能与生物对环境的适应性有关[2]。
图1 泛基因组示意图
(Annu Rev Plant Biol. 2021 Jun 17:72:411-435)[3]
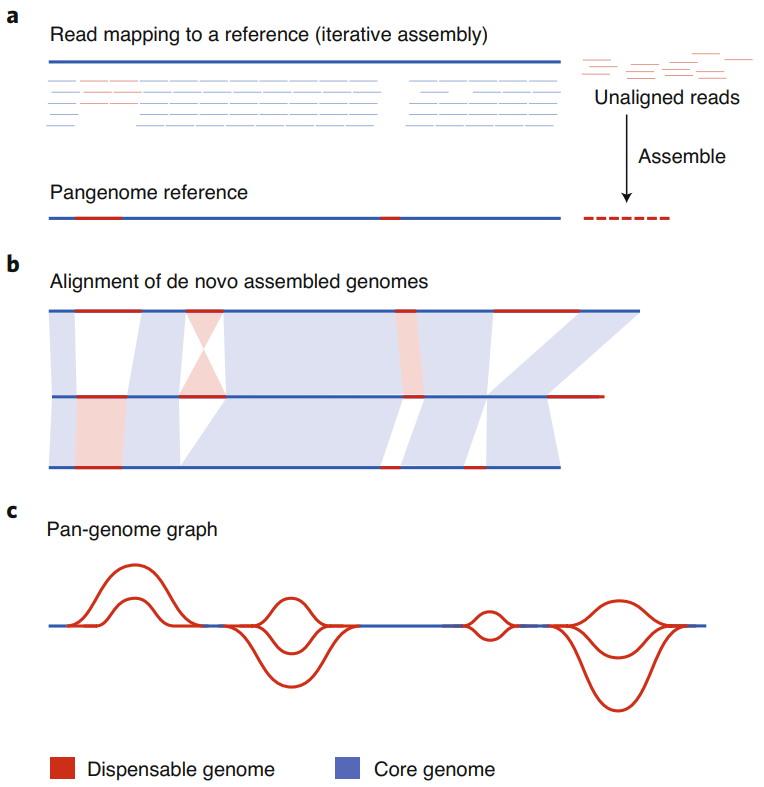
泛基因组需对多个品种/品系的基因组进行测序,然后通过多种方法整合这些基因组序列,进而形成泛基因组。泛基因组构建方法主要有3种(图2):
a:将多个样本reads逐一比对到参考基因组,将未比对上的reads组装成新的contig。通过在原始参考序列中添加这些新的contig,可以构建泛基因组参考序列。
b:多个个体基因组进行从头组装,然后通过基因组比对方法来识别可变的基因组区域。
c:图形泛基因组可以通过全基因组比对或从头组装来构建,并将可变序列以唯一路径储存在基因组中。目前,图形泛基因组是主流的构建方式。
图2 泛基因组构建方法
(Nat Plants. 2020 Aug;6(8):914-920)[4]
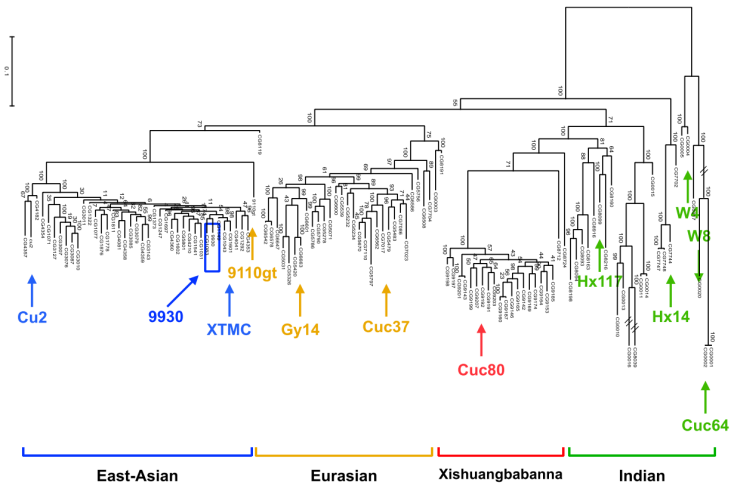
泛基因组基于多个品种/品系构建,在进行品种/品系选择时,应考虑品种\品系间的关系,亲缘关系近的材料的选择将会低估泛基因组的大小,因此要避免选择亲缘关系近的品系,如植株中可选择野生种质和栽培种质的结合的方式。同时,根据物种不同品系的聚类结果,在重要遗传分支中选择代表性品系,尽量覆盖主要的遗传变异多样性[5]。
图3 黄瓜泛基因组样品系选择
(Nat Commun. 2022 Feb 3;13(1):682)
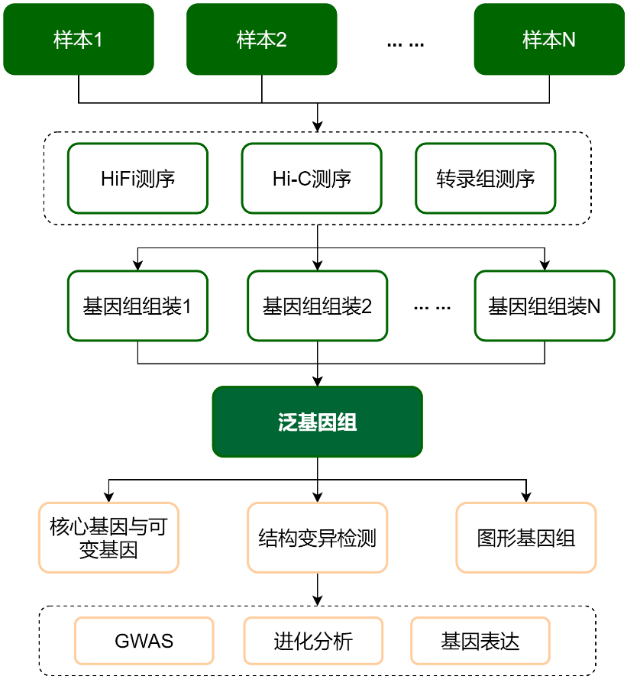
康普森基因组组装服务包括染色体级别基因组组装、动植物(泛)基因组、T2T基因组组装研究,拥有丰富的项目方案设计及分析经验,涵盖家禽、家畜、粮食作物、园艺作物、花卉林木、水产等多样性物种,将为科研工作者提供全面的三代测序、(泛)基因组和T2T基因组组装专业技术服务。
康普森泛基因组测序策略
康普森泛基因组研究方案
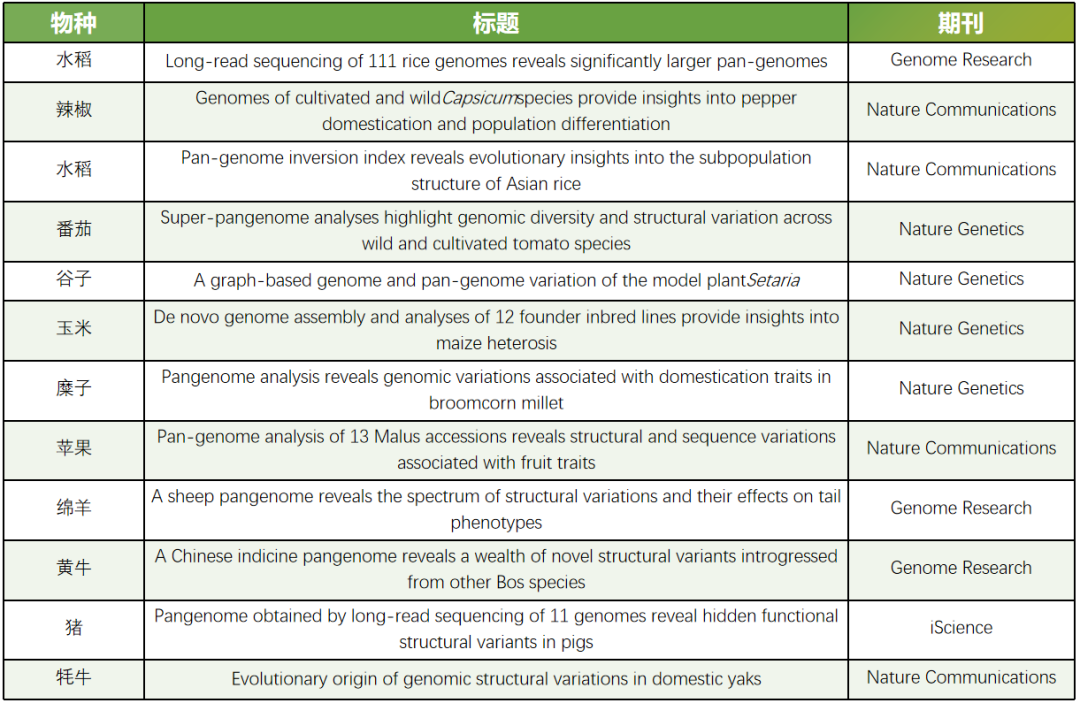
目前,泛基因组已经在植物、哺乳动物、家禽、水产、昆虫等物种的研究中获得应用,在物种功能基因组研究、进化机制、育种改良等领域都体现出重要价值,2023年以来已有多篇高水平泛基因组文章发表。
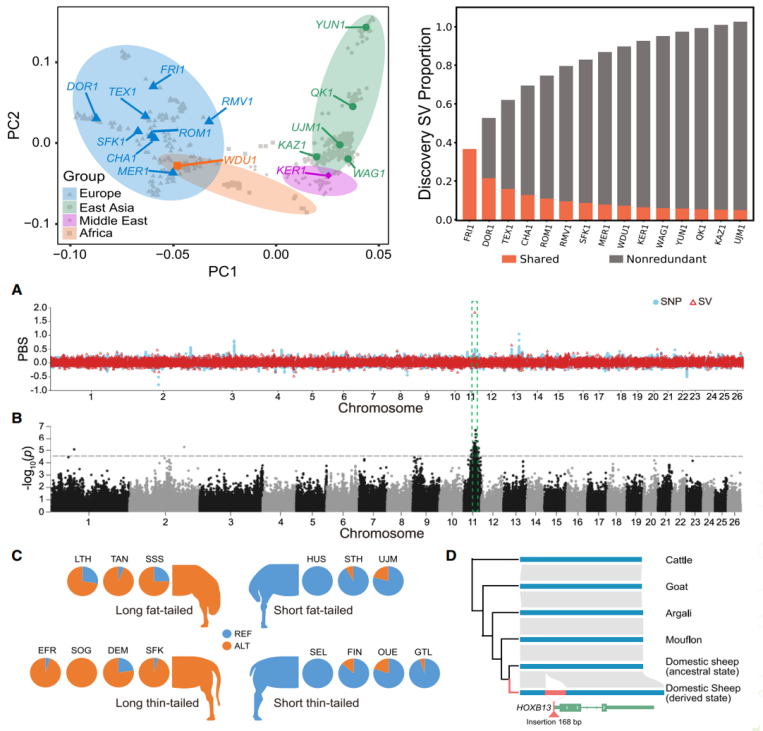
结构变异(SV)是遗传多样性和表型变异的主要原因,但在家养动物中SV的研究却少有探索。该研究使用HiFi测序对15个来自不同遗传品种的绵羊个体进行了高质量的基因组组装,并对新基因组序列和新基因进行了分析。对SV进行了精准鉴定,分析显示绵羊体内近期存在活跃的LINE扩增现象。群体进化分析确定了865个群体分层的SV,其中多个SV与不同绵羊品种的驯化有关。同时,发现HOXB13基因中插入变异与绵羊尾长性状相关。综上,该研究构建了高质量绵羊泛基因组,并鉴定了绵羊群体的SV变异集,为理解绵羊的性状生物学提供了基础资源。
图4 绵羊泛基因组及尾长性状解析
(Genome Res. 2023 Mar;33(3):463-477)
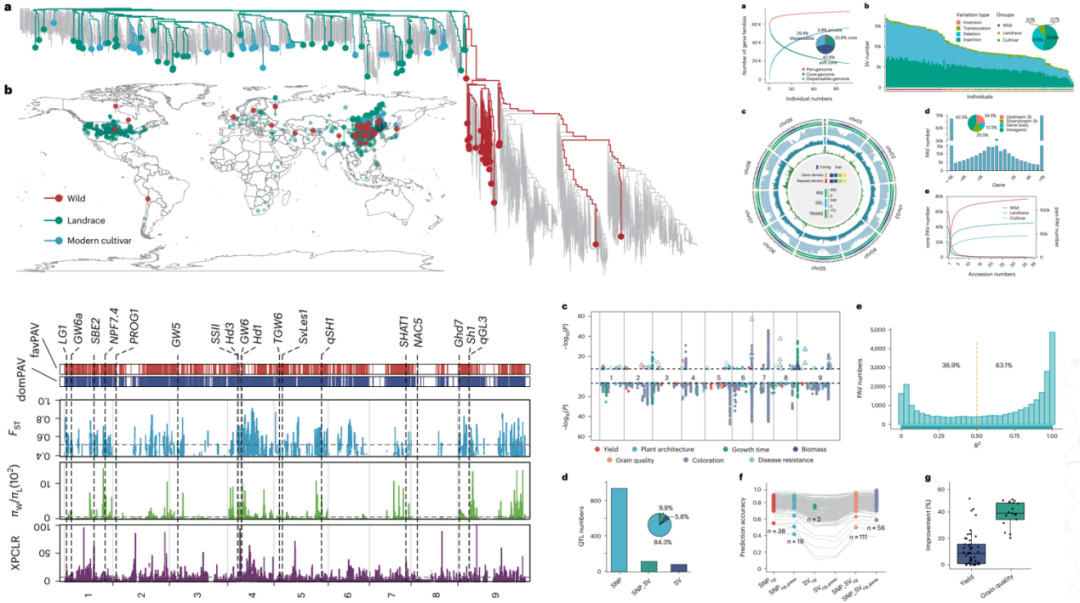
谷子是东亚的重要农作物,也是C4光合作用的模式植物。该研究通过收集来自世界各地的110个代表性基因组,建立了谷子泛基因组。泛基因组由73,528个基因家族和202,884个非冗余结构变异组成,泛基因组变异的特征表明了它们在谷子驯化和改良中的重要性。同时,基于图形泛基因组对多个性状进行了大规模的遗传研究,确定了不同地理位置里谷子品种改良的潜在基因。这些基因可用于标记辅助育种、基因组选择和基因组编辑,以加速不同气候条件下的作物改良。
图5 谷子泛基因组构建
(Nat Genet. 2023 Jul;55(7):1232-1242)
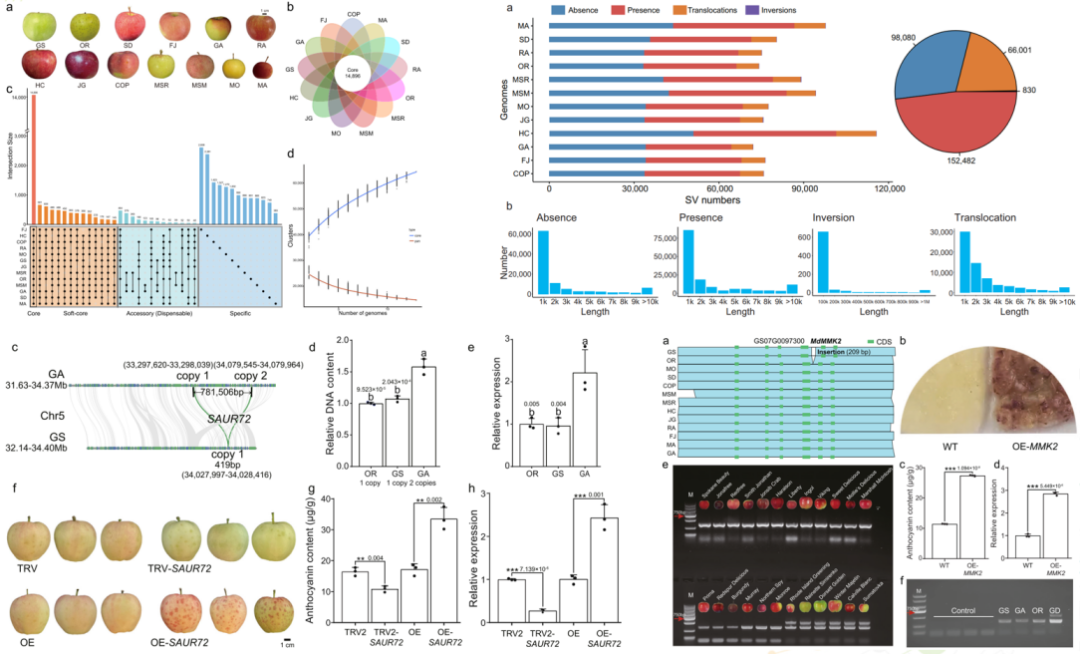
结构变异(SV)和拷贝数变异(CNV)导致了肉质结实物种的性状变异。该研究组装了10个苹果品种,结合之前报道的3个基因组,构建了苹果的泛基因组,并鉴定了20,220个CNV和317,393个SV。还观察到CNV与相关基因的表达水平呈正相关。此外,研究发现在丝裂原活化蛋白激酶同源编码基因MMK2的内含子中插入209 bp产生的非编码RNA,可以调节基因的表达并影响果实的颜色。此外,研究发现了与水果品质和生物抗性相关的重叠SV。苹果泛基因组揭示了CNV对基因表达的可能贡献,并强调了SV在苹果驯化和重要经济性状中的作用。
图6 苹果泛基因组构建
(Nat Commun. 2023 Nov 15;14(1):7377)
参考文献
赵均良, 张少红, & 刘斌. (2021). 泛基因组及其在植物功能基因组学研究中的应用. 植物遗传资源学报, 22(1), 9.
Liu, Y., Du, H., Li, P., Shen, Y., Peng, H., Liu, S., Zhou, G. A., Zhang, H., Liu, Z., Shi, M., Huang, X., Li, Y., Zhang, M., Wang, Z., Zhu, B., Han, B., Liang, C., & Tian, Z. (2020). Pan-Genome of Wild and Cultivated Soybeans. Cell, 182(1), 162–176.e13. https://doi.org/10.1016/j.cell.2020.05.023
Lei, L., Goltsman, E., Goodstein, D., Wu, G. A., Rokhsar, D. S., & Vogel, J. P. (2021). Plant Pan-Genomics Comes of Age. Annual review of plant biology, 72, 411–435. https://doi.org/10.1146/annurev-arplant-080720-105454
Bayer, P. E., Golicz, A. A., Scheben, A., Batley, J., & Edwards, D. (2020). Plant pan-genomes are the new reference. Nature plants, 6(8), 914–920. https://doi.org/10.1038/s41477-020-0733-0
Li, H., Wang, S., Chai, S., Yang, Z., Zhang, Q., Xin, H., Xu, Y., Lin, S., Chen, X., Yao, Z., Yang, Q., Fei, Z., Huang, S., & Zhang, Z. (2022). Graph-based pan-genome reveals structural and sequence variations related to agronomic traits and domestication in cucumber. Nature communications, 13(1), 682. https://doi.org/10.1038/s41467-022-28362-0
Li, R., Gong, M., Zhang, X., Wang, F., Liu, Z., Zhang, L., Yang, Q., Xu, Y., Xu, M., Zhang, H., Zhang, Y., Dai, X., Gao, Y., Zhang, Z., Fang, W., Yang, Y., Fu, W., Cao, C., Yang, P., Ghanatsaman, Z. A., … Jiang, Y. (2023). A sheep pangenome reveals the spectrum of structural variations and their effects on tail phenotypes. Genome research, 33(3), 463–477. https://doi.org/10.1101/gr.277372.122
He, Q., Tang, S., Zhi, H., Chen, J., Zhang, J., Liang, H., Alam, O., Li, H., Zhang, H., Xing, L., Li, X., Zhang, W., Wang, H., Shi, J., Du, H., Wu, H., Wang, L., Yang, P., Xing, L., Yan, H., … Diao, X. (2023). A graph-based genome and pan-genome variation of the model plant Setaria. Nature genetics, 55(7), 1232–1242. https://doi.org/10.1038/s41588-023-01423-w
Wang, T., Duan, S., Xu, C., Wang, Y., Zhang, X., Xu, X., Chen, L., Han, Z., & Wu, T. (2023). Pan-genome analysis of 13 Malus accessions reveals structural and sequence variations associated with fruit traits. Nature communications, 14(1), 7377. https://doi.org/10.1038/s41467-023-43270-7
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司








 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号