2022年4月,端粒到端粒(Telomere-to-telomere,T2T)联盟在Science公布最新的人类完整参考基因组T2T-CHM13,包括人类所有22条常染色体和X染色体的无缝组装序列,填补了过去从未组装到的~200Mb基因组序列,这些序列总长占人类基因组8%,是人类基因组史上又一重要里程碑[1]。人类T2T基因组的发布,掀起了科研工作者追求完整基因组的热潮,近一年期间,已有多个不同物种的T2T基因组相继报道,揭示基因组“黑洞”区域的遗传特征,显著提升了该物种基因组质量,对物种遗传改良、重要功能基因挖掘、遗传印记等研究起到了重要作用。
什么是T2T基因组?以高准确性的PacBio HiFi测序和高连续性的ONT ultra-long 测序为基础,组装得到的高准确性、高连续性、高完整性的端粒到端粒的高质量基因组,并对组装的端粒和着丝粒等序列进行初步验证。之所以基因组组装到T2T水平比较困难,主要是两方面的原因,一是基因组中着丝粒和端粒区域、近期片段重复、扩增基因阵列和核糖体DNA(rDNA)序列等存在高度重复区域,如水稻[2]中着丝粒含数万个155bp左右的卫星重复序列,拟南芥[3]每条染色体着丝粒含有12000-15000个178bp的串联重复,CHM13[1]基因组中含200个45k长的rDNA,香蕉[4]基因组中含110个45S rDNA。二是测序读长较短,无法跨越复杂基因组区域,造成重复序列缺失,不过,以Pacbio HiFi测序和Nanopore ultra-long测序为代表的三代测序,基于其长度长测序可以轻松跨越基因组复杂区域,使T2T组装成为可能。
康普森基因组组装服务包括染色体级别基因组组装、T2T基因组组装、动植物(泛)基因组研究,拥有丰富的项目方案设计及分析经验,涵盖家禽、家畜、粮食作物、园艺作物、花卉林木、水产等多样性物种,将为科研工作者提供全面的三代测序、(泛)基因组和T2T基因组组装专业技术服务。
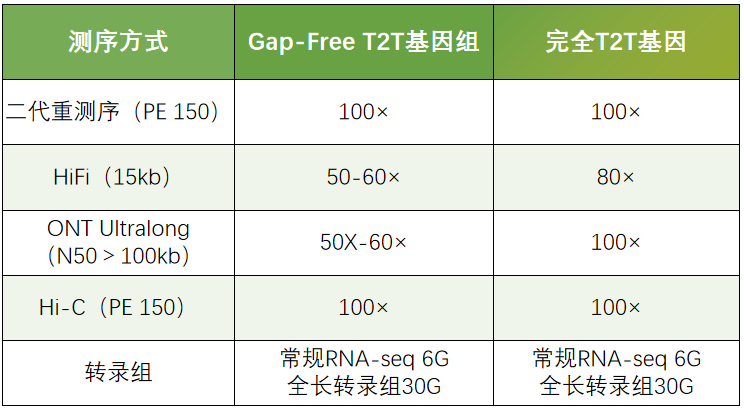
康普森T2T基因组测序策略
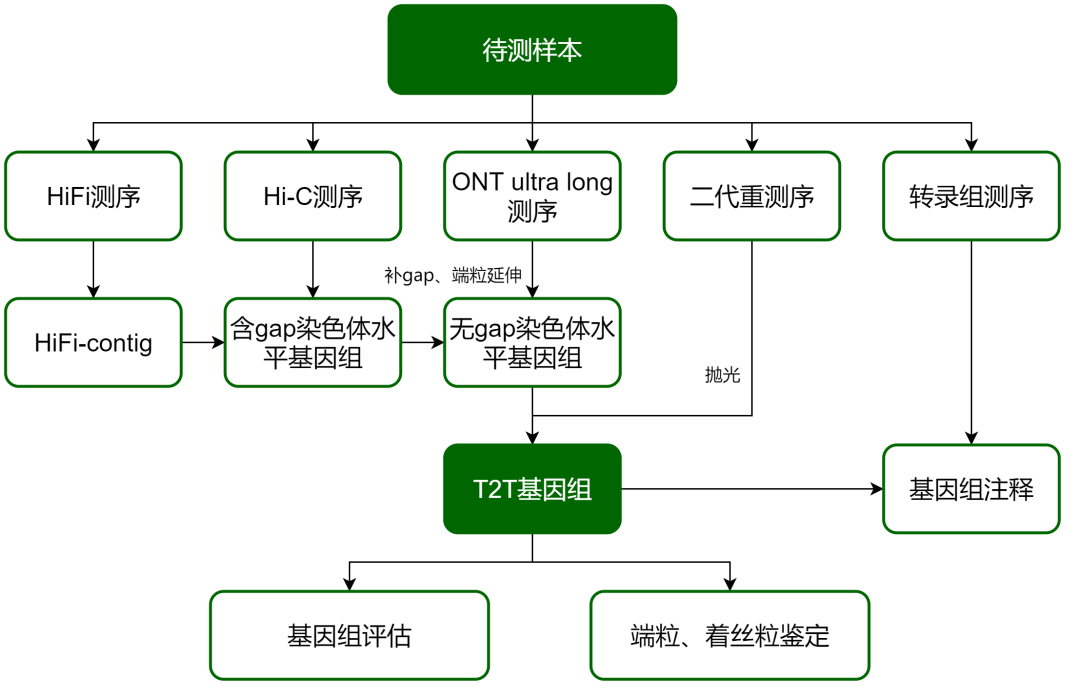
康普森T2T基因组组装策略
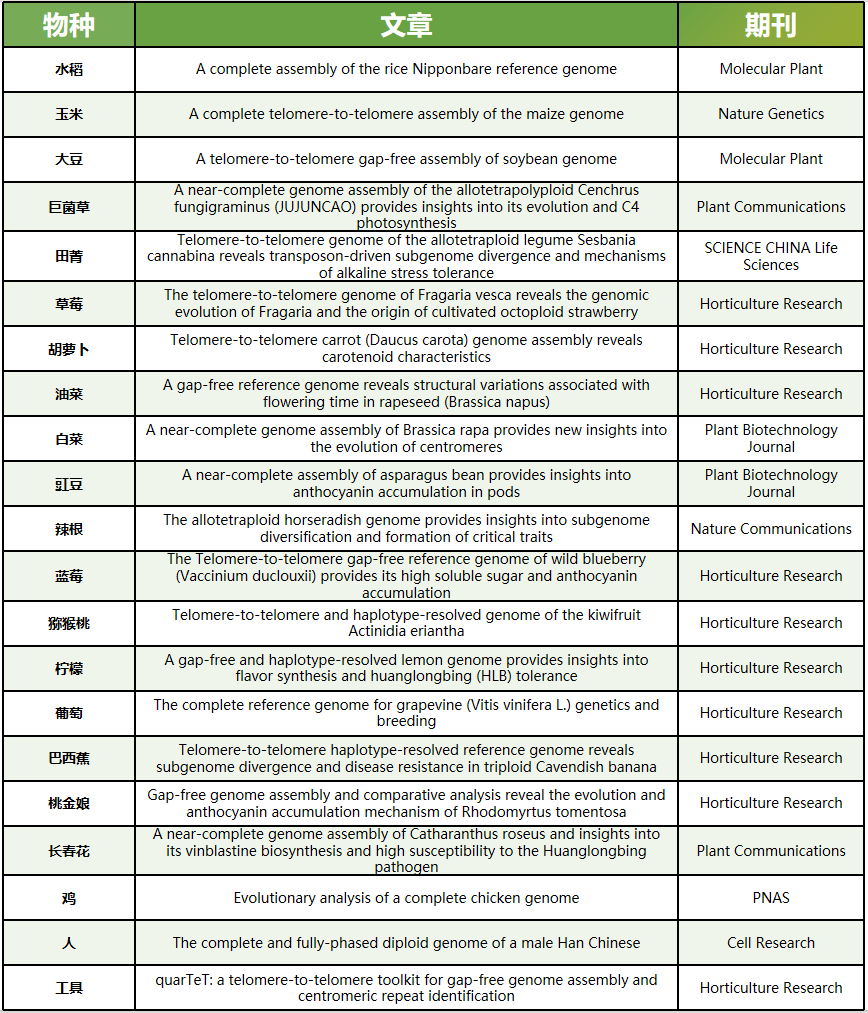
T2T基因组研究已经在人、作物、林木果树、果蔬、家禽等物种中开展,相关研究成果也受到国内外优质期刊的青睐,已有多篇高水平文章相继发表。2023年至今,已有多个物种的T2T基因组报道,编者选择多篇代表性文章分享给各位读者。
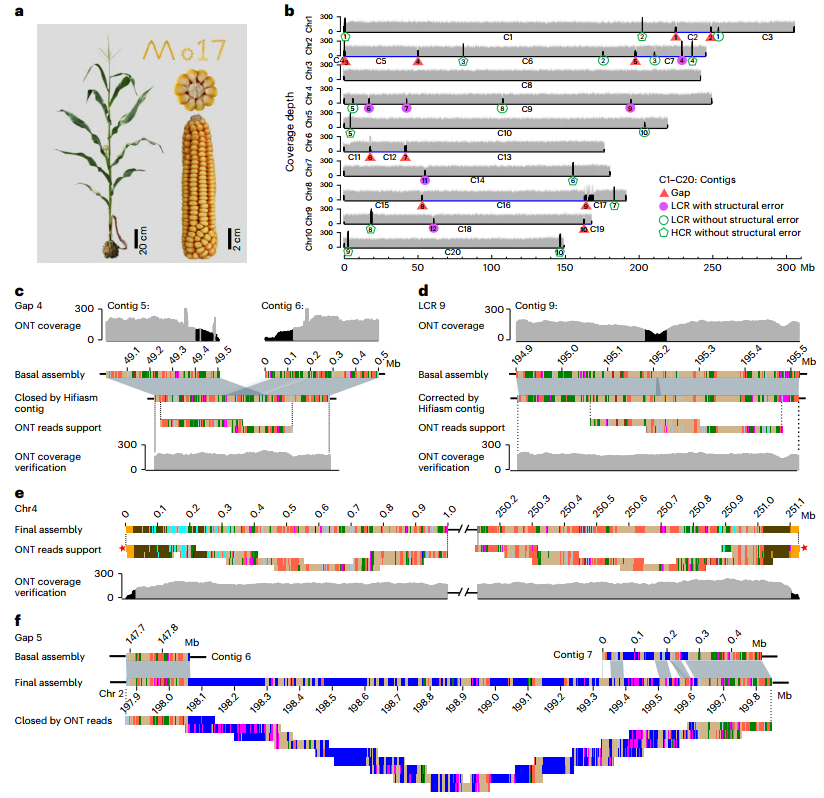
研究者通过高深度的ONT超长纳米孔测序和PacBio HiFi测序,完成了Mo17完整的基因组组装,基因组大小2178.6 Mb,碱基准确性超过99.99%,揭示了基因组所有重复区域的结构特征。有几个超长的简单重复序列阵列,其连续的TAG三核苷酸重复序列可达235 kb。在26.8 Mb的核仁组织区,包含2974个45S rDNA拷贝,显示出极其复杂的rDNA重复和转座子插入模式。此外,全部10个着丝粒的完整组装使研究者能够精确地分析富含着丝粒和缺乏着丝粒的重复序列组成。完整Mo17基因组代表着在理解高等植物基因组高度不可逆重复区域的复杂性方面迈出了重要的一步。
图1 Mo17 T2T基因组组装
(Nat Genet. 2023 Jul;55(7):1221-1231)
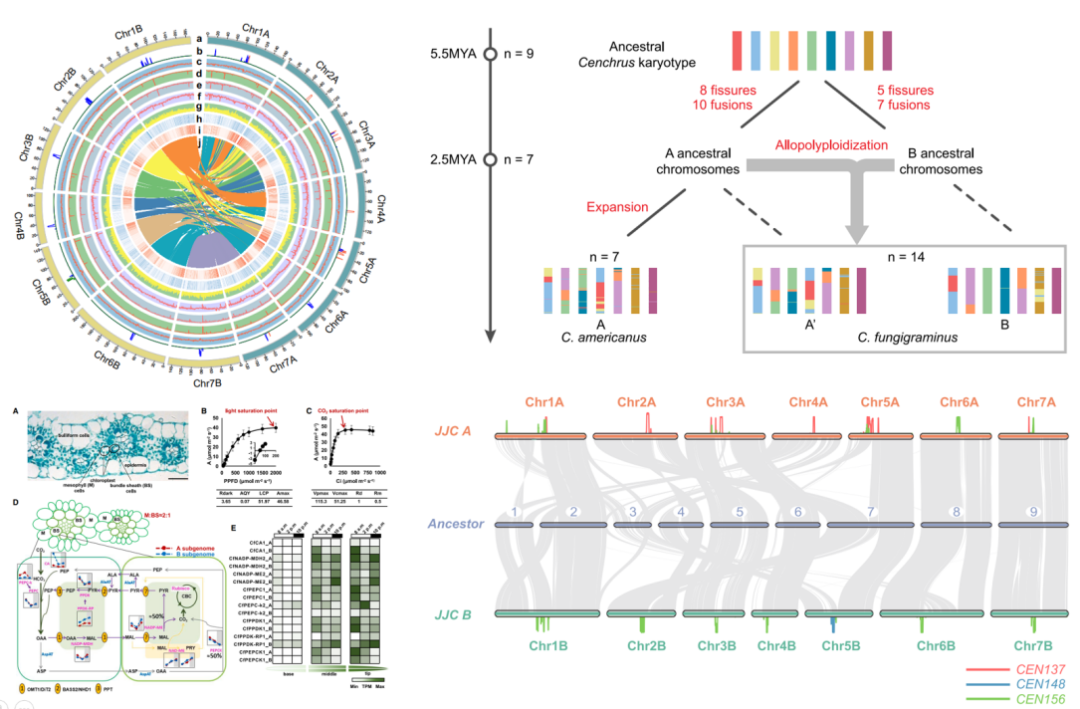
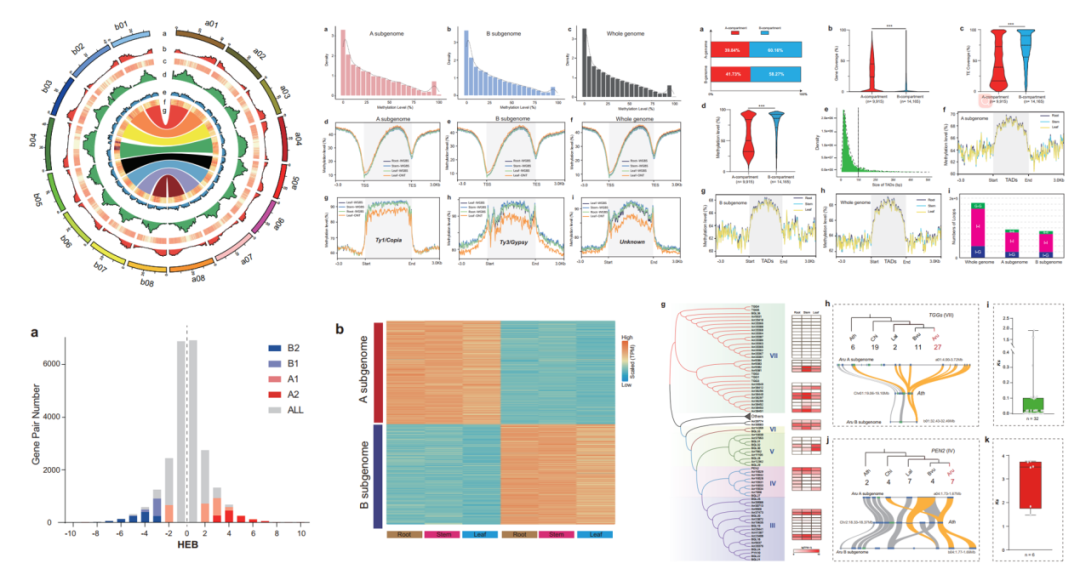
巨菌草(Cenchrus fungigraminus)是栽培植物中生物量产量最高的生物,可用于蘑菇栽培、动物饲料和生物燃料生产。该研究报告了巨菌草几乎完整的基因组组装,并揭示了巨菌草是起源于270万年前的异源多倍体。其基因组由两个亚基因组组成,A亚基因组与珍珠稷具有高度共线性。巨君草的基因组进化显示蒺藜草属的祖先核型分化为巨君草的A和B祖先核型。比较转录组和DNA甲基化组分析显示两个亚基因组之间同源基因对的功能分化,进一步表明DNA不对称的甲基化。三种着丝粒重复序列(CEN137, CEN148和CEN156)可能在每个亚基因组中独立进化。通过对巨菌草光合特性的研究,揭示了其典型的C4 Kranz结构和较高的光合效率,与其他C4光合亚型不同的是,巨菌草在主要的C4脱羧反应中存在NADP-ME和PEPCK的协同作用,这可能有助于菊花草获得较高的光合效率和生物量。
图2 巨菌草 T2T基因组特征
(Plant Commun. 2023 Sep 11;4(5):100633)
多倍体化可以为适应性进化和物种形成提供丰富的遗传变异,但对亚基因组进化的及其动力学机制的理解仍难以捉摸的。该研究使用多种测序策略,开展异源四倍体辣根T2T无间隙参考基因组组装。A和B亚基因组的基因组结构、表观遗传结构和三维染色质结构差异显著,表明显性长末端重复反转录转座子的动力学特性和DNA甲基化在亚基因组分化中发挥了关键作用。对硫代葡萄糖苷和辣根过氧化物酶生物合成的遗传基础的研究揭示了多倍体化和亚基因组分化在重要性状建成中的重要作用。硫代葡萄糖苷生物合成必需基因(如FMOGS-OX、IGMT和GH1基因家族)的持续重复和分化有助于辣根广泛的硫代葡萄糖苷分布。总之,异源四倍体辣根基因组的T2T组装扩展了对多倍体基因组进化的认识,为辣根的育种和遗传改良提供了基础遗传资源。
图3 辣根基因组特征、亚基因组差异及硫代葡萄糖苷合成
(Nat Commun 2023 Jul 25;14(1):4102)
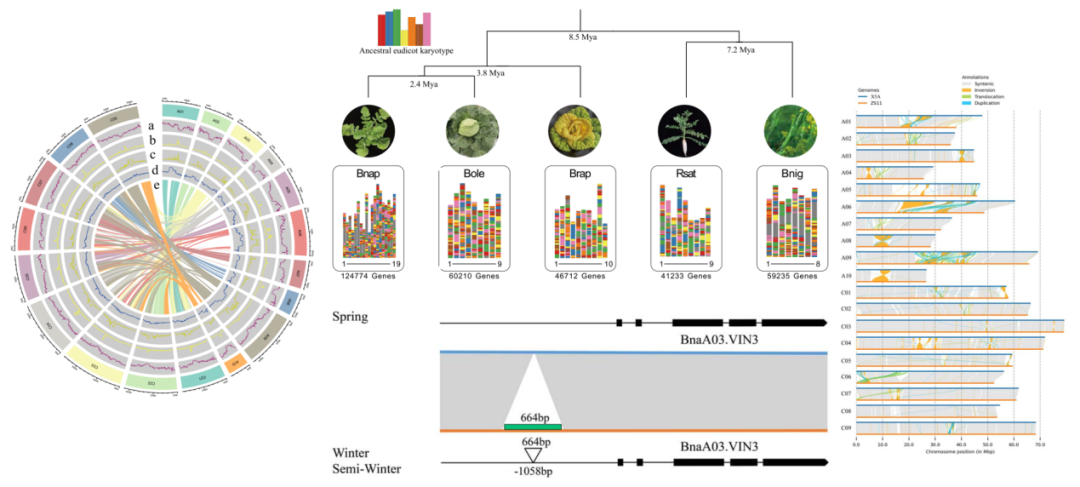
油菜是重要的油料作物和蔬菜作物,该研究报告了一个油菜品种湘5A的无间隙基因组,使用ONT超长reads、PacBio HiFi reads和Hi-C数据,组装获得全部19条无间隙染色体,其中8条染色体是T2T组装。与已发表的油菜基因组相比,无间隙基因组在无人工干预情况下,完整组装了19条染色体中的9条,大大提高了基因组的连续性和完整性,代表了迄今为止最高质量的基因组组装。分析结果表明,湘5A经历了完整的三倍化和异位四倍化。通过无间隙基因组,发现917个开花相关基因受到了结构变异的影响,包括BnaA03.VERNALIZATION INSENSITIVE 3和BnaC04.HIGH EXPRESSION OF OSMOTICALLY RESPONSIVE GENES 1,这些基因可能在调节湘5A开花时间和促进湘5A在长江流域的适应中发挥重要作用。该无间隙基因组为油菜功能基因组研究和育种提供了宝贵的遗传资源。
图4 油菜基因组特征、基因组进化及结构变异
(Hortic Res. 2023 Aug 29;10(10):uhad171)
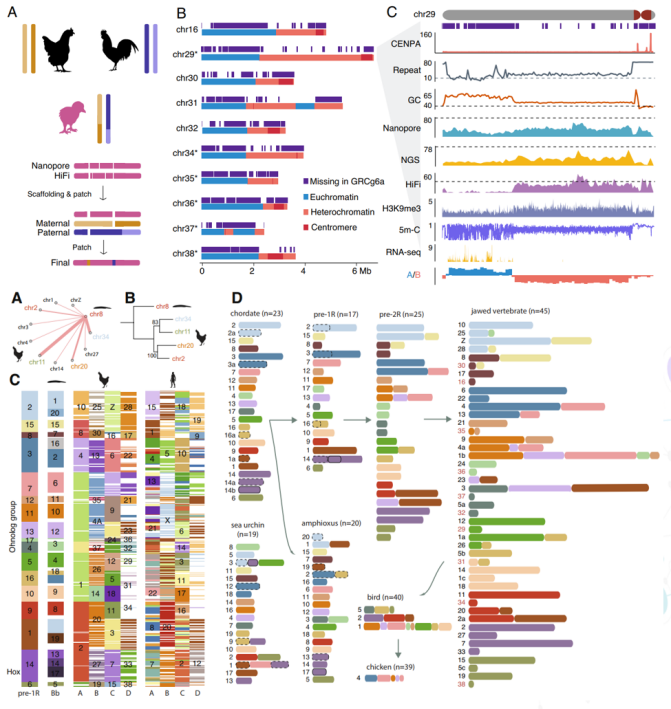
该研究对1个“胡须”品种的母鸡进行三代测序,首先分别使用ONT和HiFi reads进行基因组组装,然后HiFi组装contig替换ONT组装contig并进行GAP填充,获得T2T基因组GGswu1。同时结合trio-sequencing的方法组装得到了2个单倍型基因组。研究鉴定了多条新的点染色体,并分析了点染色体、着丝粒、端粒的序列结构特征。同时,重建了脊索动物核型进化的精细图像,揭示了脊椎动物全基因组重复前后频繁的染色体融合。该研究对鸡染色体的序列和表观遗传特征的分析有助于理解脊椎动物的基因组进化和染色体生物学。
图5 家鸡T2T基因组组装及核型进化分析
(Proc Natl Acad Sci USA. 2023 Feb 1;120(8):e2216641120)
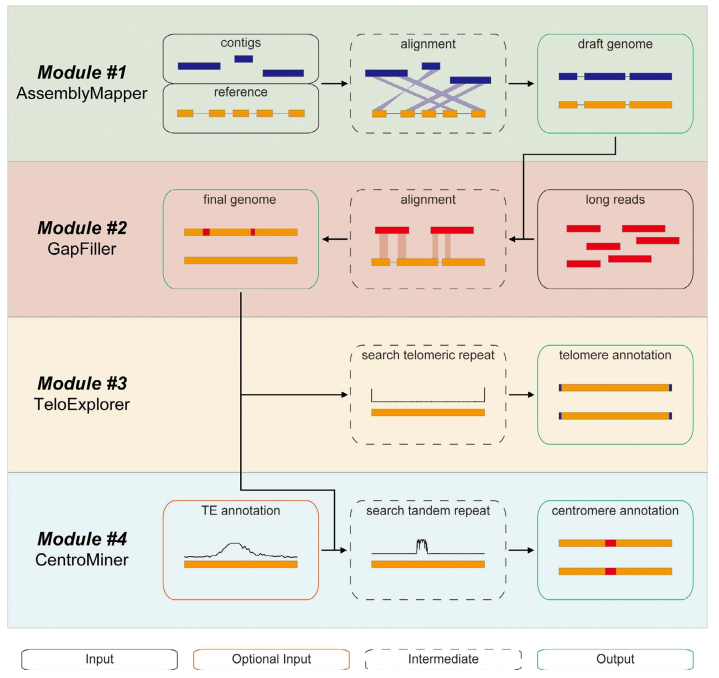
开发了一个专门为T2T基因组组装和表征设计的用户友好的网络工具quarTeT,目前包括四个模块:AssemblyMapper, GapFiller, TeloExplorer和CentroMiner。模块一AssemblyMapper基于参考基因组将contigs组装到染色体水平,模块二GapFiller利用long reads填补染色体gap,模块三TeloExplorer鉴定端粒结构特征,模块四CentroMiner鉴定着丝粒结构特征。这四个模块可以单独使用或相互组合用于T2T基因组组装和表征。研究者采用了quarTeT的全部模块化功能,实现了猕猴桃基因组的T2T组装,并使用CentroMiner评估了拟南芥和水稻基因组中着丝粒区域。总之,quarTeT是研究大规模T2T基因组的有效工具包。
图6 quarTeT分析流程
(Hortic Res. 2023 Jun 13;10(8):uhad127)
总之,随着测序读长的增加、测序质量的提升、测序成本的降低以及基因组分析方法的优化,T2T基因组组装已经不再是遥不可及,并且会成为今后发展的必然趋势,照亮基因组“黑洞”,解析复杂基因组结构,完整展现物种遗传信息,T2T基因组技术将会打开生物学研究的新局面。
2023年T2T基因组文章汇总
参考文献
【1】Nurk S, Koren S, Rhie A, et al. The complete sequence of a human genome. Science. 2022;376(6588):44-53. doi:10.1126/science.abj6987
【2】Song JM, Xie WZ, Wang S, et al. Two gap-free reference genomes and a global view of the centromere architecture in rice. Mol Plant. 2021;14(10):1757-1767.doi:10.1016/j.molp.2021.06.018
【3】Naish M, Alonge M, Wlodzimierz P, et al. The genetic and epigenetic landscape of the Arabidopsis centromeres. Science. 2021;374(6569):eabi7489. doi:10.1126/science.abi7489
【4】Chen J, Wang Z, Tan K, et al. A complete telomere-to-telomere assembly of the maize genome. Nat Genet. 2023;55(7):1221-1231. doi:10.1038/s41588-023-01419-6
【5】Zheng H, Wang B, Hua X, et al. A near-complete genome assembly of the allotetrapolyploid Cenchrus fungigraminus (JUJUNCAO) provides insights into its evolution and C4 photosynthesis. Plant Commun. 2023;4(5):100633. doi:10.1016/j.xplc.2023.100633
【6】Shen F, Xu S, Shen Q, Bi C, Lysak MA. The allotetraploid horseradish genome provides insights into subgenome diversification and formation of critical traits. Nat Commun. 2023;14(1):4102. Published 2023 Jul 25. doi:10.1038/s41467-023-39800-y
【7】Li B, Yang Q, Yang L, et al. A gap-free reference genome reveals structural variations associated with flowering time in rapeseed (Brassica napus). Hortic Res. 2023;10(10):uhad171. Published 2023 Aug 29. doi:10.1093/hr/uhad171
【8】Huang Z, Xu Z, Bai H, et al. Evolutionary analysis of a complete chicken genome. Proc Natl Acad Sci U S A. 2023;120(8):e2216641120. doi:10.1073/pnas.2216641120
【9】Lin Y, Ye C, Li X, et al. quarTeT: a telomere-to-telomere toolkit for gap-free genome assembly and centromeric repeat identification. Hortic Res. 2023;10(8):uhad127. Published 2023 Jun 13. doi:10.1093/hr/uhad127
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司











 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号