QTL定位是找到与目标性状相关基因的经典研究方法之一,其原理是基于分子标记与目标性状的连锁关系,找到与目标性状相关区域。
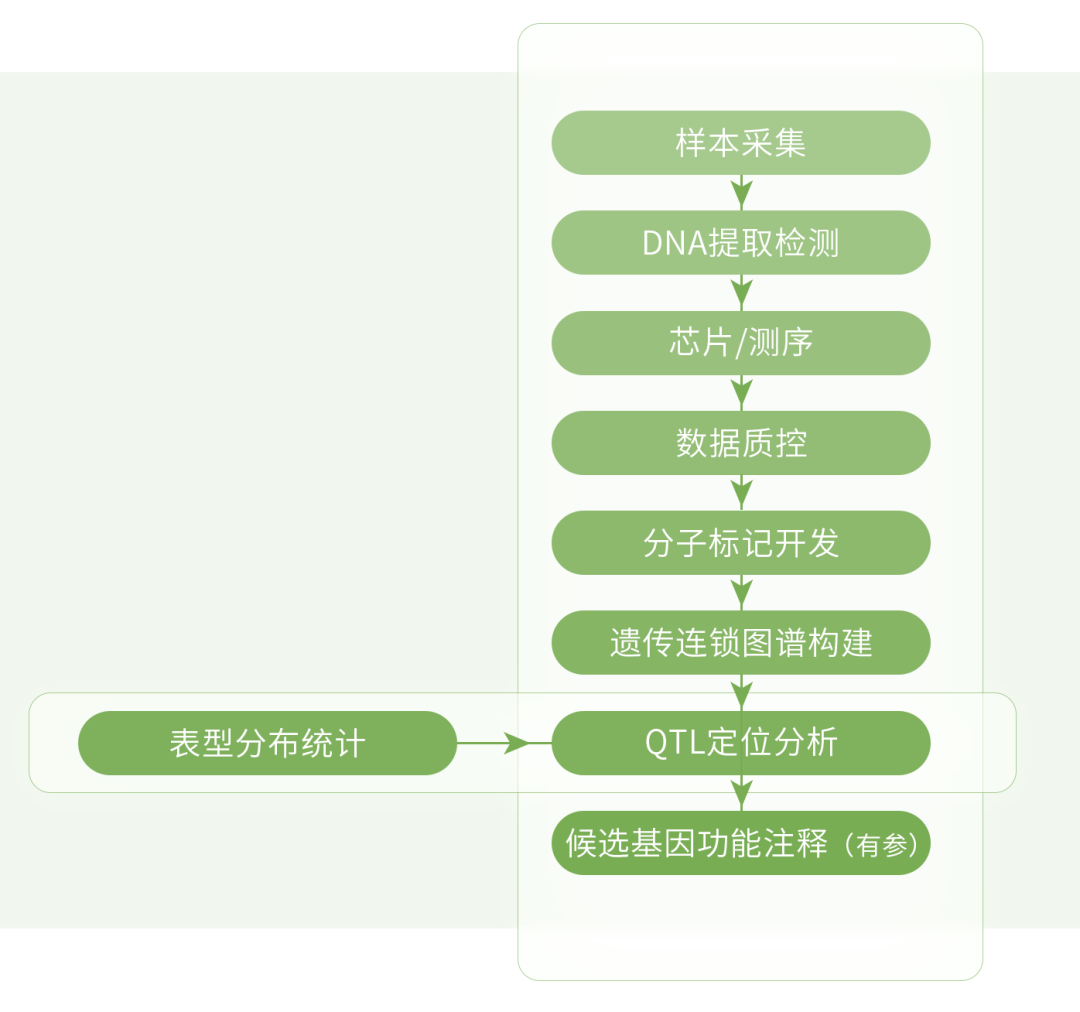
上篇分享了关于QTL定位相关原理、常见作图群体类型、分析流程以及常见结果可视化。本篇着重介绍康普森作物遗传图谱构建及QTL定位产品,同时针对QTL定位常见问题进行解答。
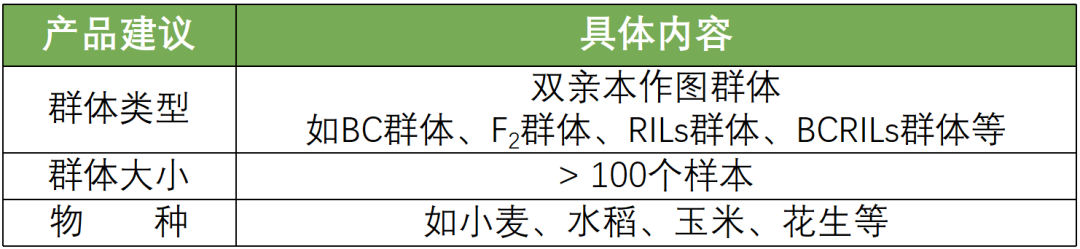
康普森作物遗传图谱构建及QTL定位产品,涵盖了表型统计、分子标记开发、遗传图谱构建、连锁分析、QTL区间位点注释、区间基因功能注释及富集分析的内容。客户可根据需求自行选择遗传图谱构建产品、QTL定位(需提供表型数据)产品,具体产品适用性见下表。
表型数据统计
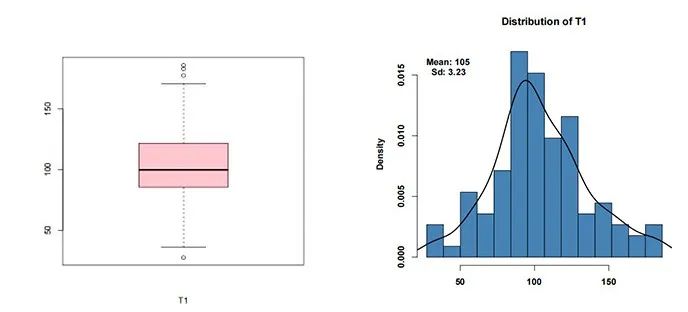
表型对QTL定位至关重要,一般要求表型数据的随机误差服从正态分布。分析前对作图个体表型数据绘制箱线图,将严重偏离总体分布的个体表型替换为NA,而后绘制表型分布直方图,如图1所示。
图1 性状N表型分布箱线图与直方图
分子标记开发
对基因型及表型进行一定标准的质控,可以排除异常基因型和异常表型对分析结果产生的影响。
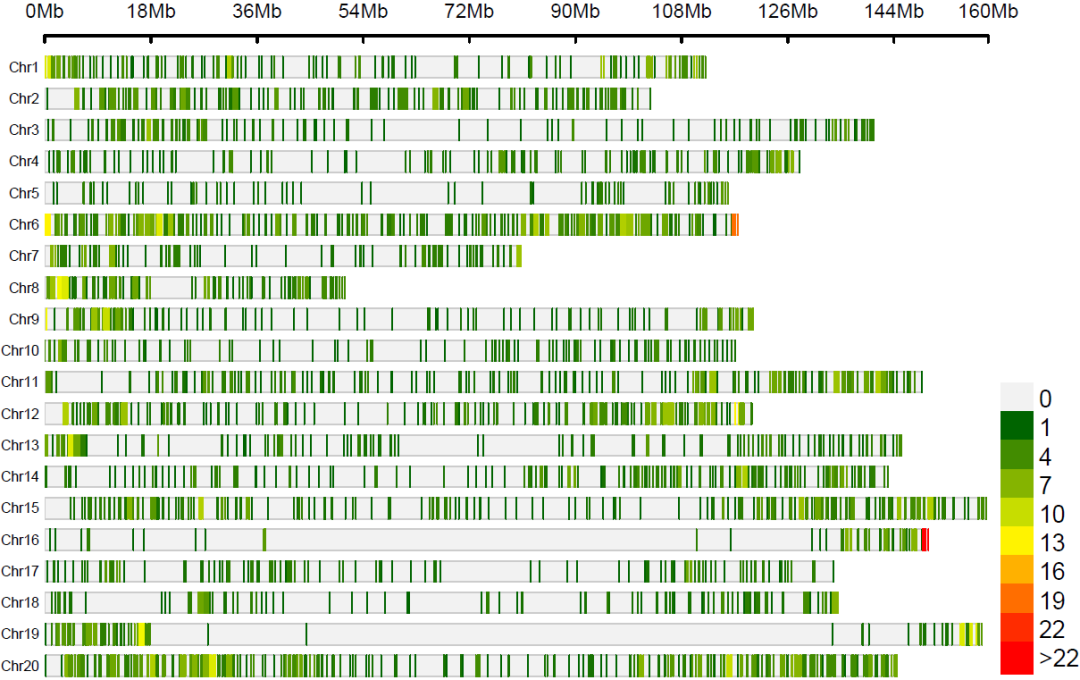
QTL定位分析前,首先筛选两个亲本均为纯合基因型且亲本间差异的标记位点,然后对子代群体数据进行基因型质控。基因型质控含SNP检出率、最小等位基因频率、标记偏分离卡方检验等。若为RIL群体等,则会根据个体杂合率、位点杂合率剔除部分杂合个体、杂合位点,质控后剩余标记的密度分布请见图2。
图2 筛选用于遗传图谱构建的标记密度分布示例图
遗传图谱构建
对筛选得到的高质量标记,采用QTL IciMapping (Version 4.2) 软件进行遗传图谱构建,然后统计遗传图谱信息,见附件报告模板“4.遗传图谱构建”。
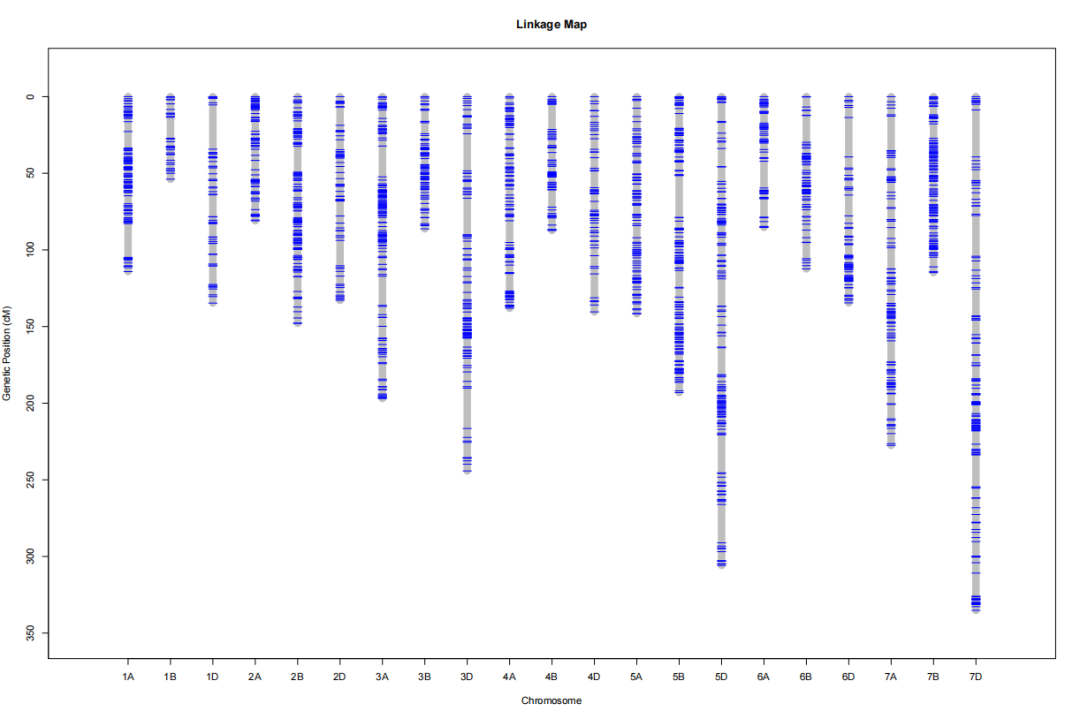
图3 遗传图谱标记分布示例图
QTL定位分析
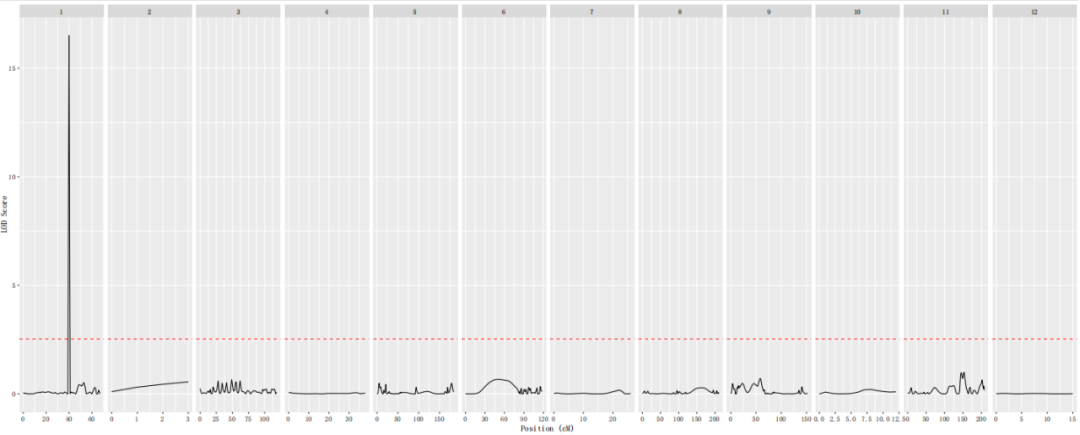
控制数量性状的基因在基因组中的位置称为数量性状基因座(Quantitative Trait Locus,QTL)。QTL 常用作图方法、软件较多,康普森分析采用QTL IciMapping (Version 4.2) 软件基于完备区间作图法(ICIM)进行QTL定位。针对客户提供的表型数据,统计所有性状鉴定到的QTLs,QTL定位结果示例见报告模板“5.QTL定位分析”,QTL 定位结果示例展示见图4。
图4 性状N QTL定位结果展示
注:纵坐标为 LOD 值;红色的线为LOD阈值线;上横坐标为
不同连锁群编号;下横坐标为每条连锁群的遗传距离。
QTL 区间注释
基于 QTL 定位结果,筛选出与性状关联的 SNP 位点,进行区间位点注释。基于ANNOVAR位点注释结果,使用clusterProfiler包将注释到的区间候选基因,根据GO和KEGG数据库进行基因功能富集分析。
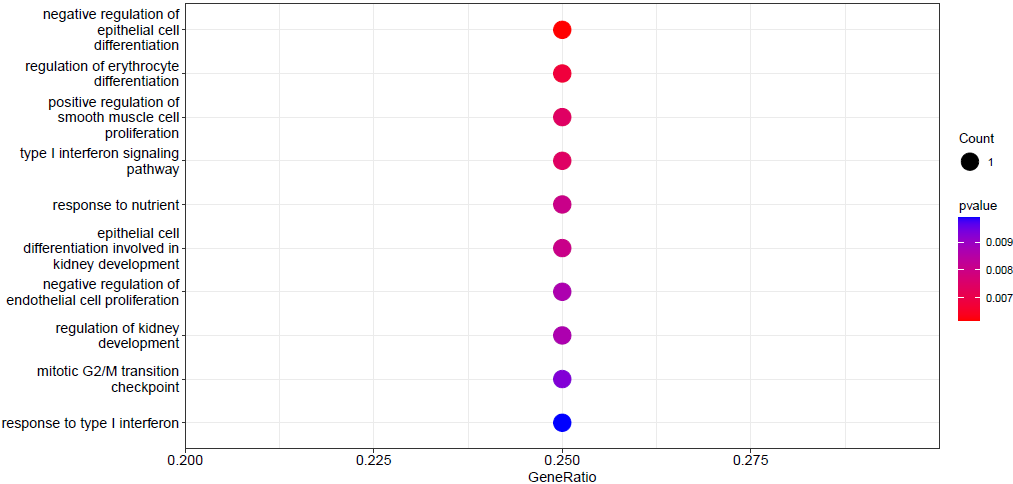
基于连锁关系,QTL定位到的区间往往包含较多候选基因,客户可自行参考富集分析结果,进一步筛选与目标性状相关候选基因,GO富集分析的结果示例如下图5所示。
图5 GO biological process(BP)ontology富集分析结果点状图
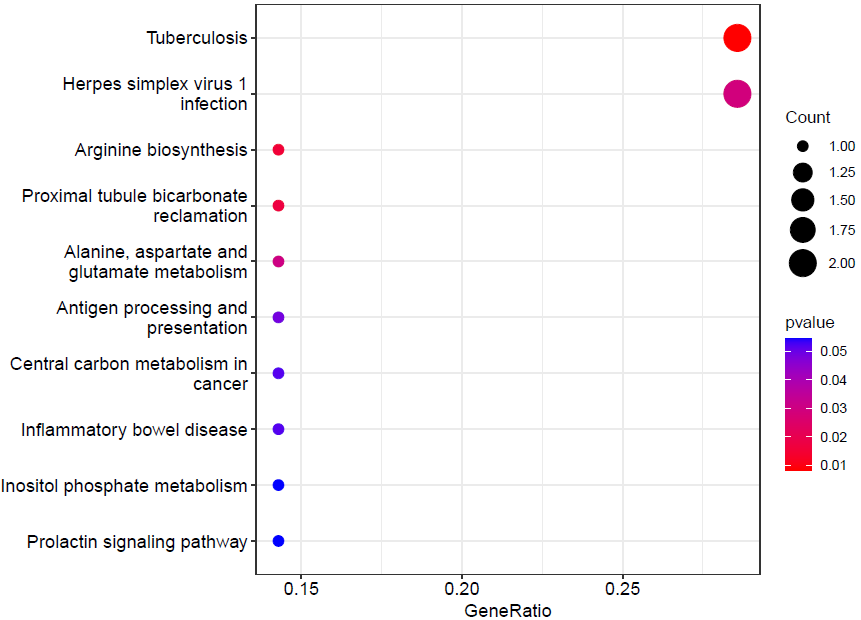
KEGG 是有关通路的主要公共数据库,通路显著性富集分析以 KEGG 中的通路为单位。应用超几何检验,找出与整个参考基因相比较在候选基因中显著性富集的通路,KEGG通路分析结果示例如下图6所示。
图6 KEGG通路分析结果点状图
作物遗传图谱构建、QTL定位产品区别
图谱构建是QTL定位的基础,提供亲本及子代测序/芯片数据则可进行遗传图谱构建,除此之外,提供群体目标表型数据则可进一步进行QTL定位,找到与目标性状相关的QTLs。
群体构建亲本的选择
亲本的选择、群体构建方式影响着最终的定位结果。亲本的选择需要综合考虑亲本间遗传背景差异、纯度、杂交后代可育性(偏分离)等因素。
如何提高 QTL 检测功效
QTL定位是一种统计方法,QTL分析假设性状值的随机误差项服从正态分布。检测功效与准确性受目标性状的可遗传性、测量准确性、群体类型、群体大小、定位方法、标记数量等的影响。一些QTL可能只在特定杂交群体,或是特定环境下才可以被检测到的。
提高 QTL 的检测功效主要途径:1. 扩大作图群体,即在允许条件下要尽可能地扩大作图群体,增加重组事件。考虑到构建成本,已有研究大多子代数200个个体左右;2. 减小表型误差,如可以通过统一性状测量标准,多地点、多年份采集表型等方法提高QTL检测的可信度;3. 适当增加标记密度,已有研究表明在低于200的作图群体中, 标记增加到一定程度, 如每隔5~10 cM有一个标记, 再增加更多的标记也不会明显提高QTL的检测功效。
参考文献
[1]Wang, J, H. Li, L. Zhang, and L. Meng. 2019. Users’Manual of QTL IciMapping. The Quantitative Genetics Group, Institute of Crop Science, Chinese Academy of Agricultural Sciences (CAAS), Beijing 100081, China.
[2]李慧慧,张鲁燕,王建康.数量性状基因定位研究中若干常见问题的分析与解答[J].作物学报,2010,36(06):918-931.
[3]Wang J-K(王建康). Inclusive composite interval mapping of quantitative trait genes. Acta Agron Sin (作物学报), 2009, 35(2): 239–245 (in Chinese with English abstract).
[4]Purcell S, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet., 2007. 81(3): 559–75.
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司









 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号